Reconnaître des oiseaux grâce à leur chant
Biodiversité aviaire dans les Ghâts occidentaux, en Inde.
Compétion Kaggle BirdCLEF 2024 https://www.kaggle.com/competitions/birdclef-2024/overview
Les oiseaux sont d'excellents indicateurs de changement de la biodiversité car ils sont très mobiles et ont des exigences d'habitat diverses. Les changements dans l'organisation des espèces et le nombre d'oiseaux peuvent donc indiquer le succès ou l'échec d'un projet de restauration. Cependant, réaliser fréquemment des enquêtes traditionnelles basées sur l'observation de la biodiversité aviaire sur de grandes surfaces est coûteux et logistiquement difficile. En comparaison, la surveillance acoustique passive (PAM) combinée à de nouveaux outils analytiques basés sur l'apprentissage automatique permet aux conservationnistes d'échantillonner des échelles spatiales beaucoup plus grandes avec une résolution temporelle plus élevée et d'explorer en profondeur la relation entre les interventions de restauration et la biodiversité.
On va développer ici une solution informatique pour traiter les données audio continues et reconnaître les espèces par leurs chants. Notre objectif est d'aider à faire progresser les efforts en cours pour protéger la biodiversité aviaire dans les Ghâts occidentaux, en Inde.

Contexte¶

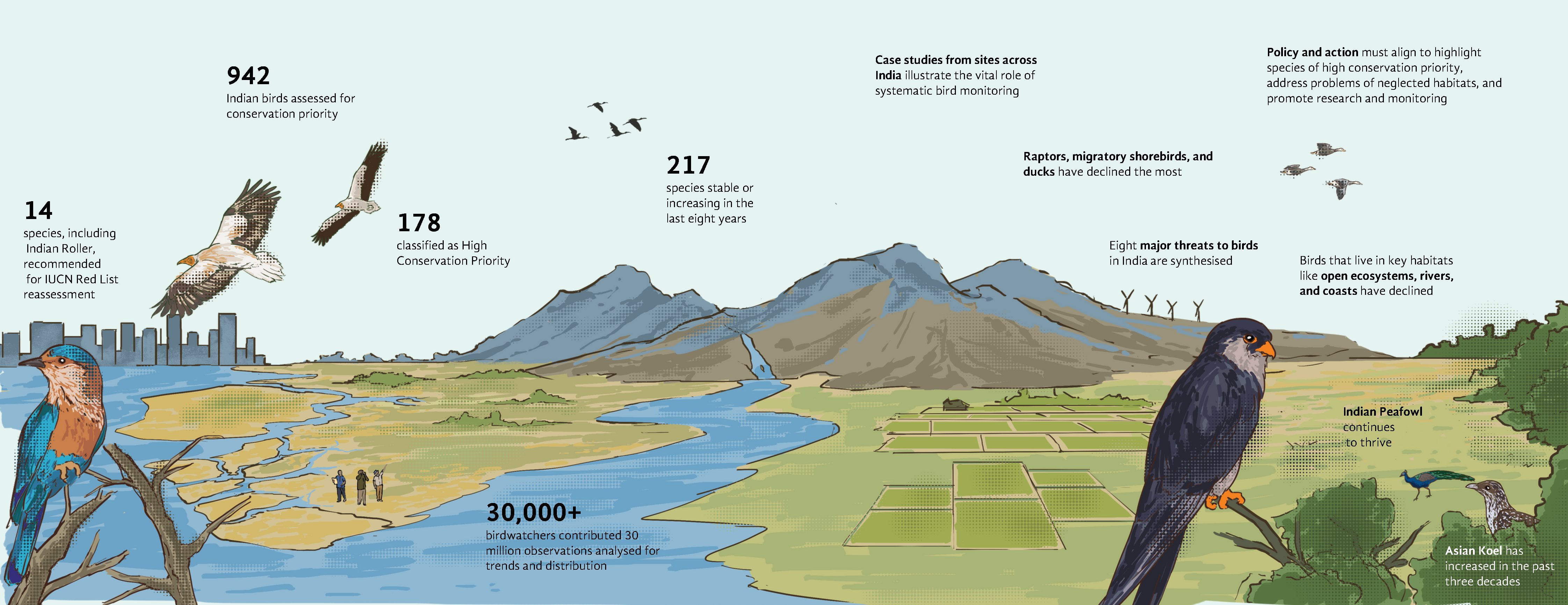
Cette page nous permettra d'effectuer d'éventuelles vérifications : https://stateofindiasbirds.in/species/trends-in/ca-none/habitats-none/estimated_trend-yes/
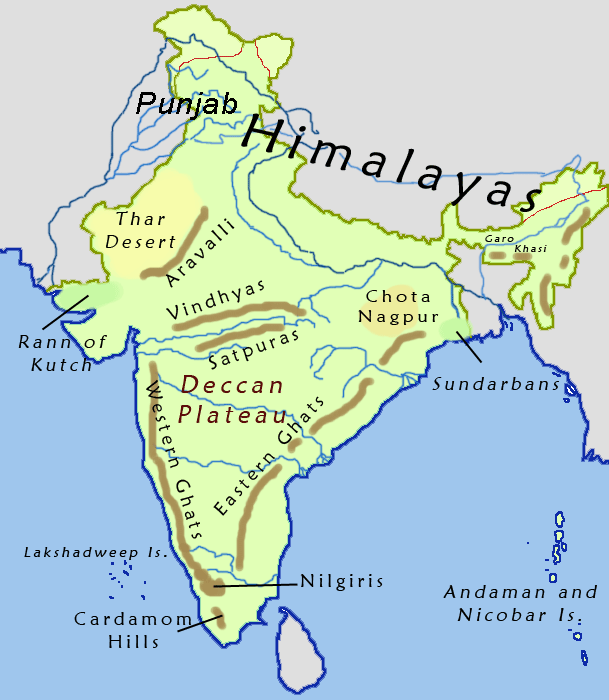
Les Ghâts occidentaux¶
De plus, cette région abrite de grandes populations humaines dont les moyens de subsistance dépendent des forêts et des ressources naturelles qu'elle offre. D'un point de vue avifaune, cette région abrite des niveaux élevés de diversité des oiseaux, avec plusieurs espèces endémiques et menacées qu'on ne trouve nulle part ailleurs. Cependant, cette chaîne de montagnes subit des changements drastiques de paysage et de climat qui affectent négativement la biodiversité. D'où le besoin de technologies et d'outils de conservation pour aider à évaluer et à surveiller rapidement la diversité des oiseaux.
Dès 2017 les ornithologues s'inquiètaient de la fragiliié de la région. Ici (https://evolecol.weebly.com/blog/2017-western-ghats-birds) par exemple, on traite des oiseaux endémiques des Ghats occidentaux et de la façon dont ils sont plus menacés qu'on ne le pensait auparavant. Les cartes de répartition utilisées par l'Union internationale pour la conservation de la nature (UICN) pour ces espèces sont inexactes et surestiment la taille de la zone que les oiseaux habitent réellement.
Une étude a été menée en utilisant des données de eBird et d'autres sources pour créer des cartes de répartition plus précises pour 18 espèces d'oiseaux endémiques des Ghats occidentaux. Les résultats ont montré que les cartes de répartition utilisées par l'UICN surestiment considérablement la taille de la zone habitée par ces oiseaux.
Les auteurs appellent à une approche plus rigoureuse pour déterminer les gammes d'espèces et à une meilleure prise en compte des zones de conservation de grande valeur.
Nous nous inspirerons de cette étude pour établir la méthode de détection algorithmique des oiseaux en danger et nous retenons la liste des 18 oiseaux endémiques cités à savoir :
- Nilgiri pipit
- Wynaad laughingthrush
- Malabar grey hornbill
- Rufous babbler
- Grey-fronted green pigeon
- White-bellied treepie
- Grey-headed bulbul
- Malabar parakeet
- Nilgiri wood pigeon
- Nilgiri flycatcher
- Broad-tailed grassbird
- Black-and-orange flycatcher
- Black-chinned laughingthrush
- Crimson-backed sunbird
- White-bellied shortwing
- Nilgiri shortwing
- White-bellied blue flycatcher
- Kerala laughingthrush
Elle nous servira de repère de la qualité de notre travail.
Objectifs du projet¶
En préambule nous annoncions l'objectif général, voici les objectifs secondaires et techniques. Il nous faudra avoir en tête de :
(1) Identifier les espèces d'oiseaux endémiques des îles célestes des Ghâts occidentaux dans les données de paysage sonore.
(2) Détecter/classifier les espèces d'oiseaux en danger (espèces préoccupantes pour la conservation) avec des données d'entraînement limitées.
(3) Détecter/classifier les espèces d'oiseaux nocturnes qui sont mal comprises.
Nous allons construire un outil à l'aide de données d'entraînement limitées. Il pourra se mettre, bien entendu, à jour avec des données nettement plus consistantes. Cet état de fait devra également relativiser également certaines frustrations face à des résultats semblant décévant.
Le modèle de machine learning utilisera comme métrique d'évaluation une version de la ROC-AUC moyenne macro qui exclut les classes n'ayant pas d'étiquettes positives vraies. https://www.linkedin.com/advice/1/what-best-practices-handling-roc-auc-score-predictive-qvxkc?lang=fr&originalSubdomain=fr
Fichiers en présence¶
- train_audio/
Les données d'entraînement se composent de courts enregistrements de cris d'oiseaux individuels généreusement téléchargés par des utilisateurs de xenocanto.org. Ces fichiers ont été rééchantillonnés à 32 kHz, le cas échéant, pour correspondre à l'audio de l'ensemble de test et convertis au format ogg. Les données d'entraînement devraient avoir presque tous les fichiers pertinents ; nous pensons qu'il n'est pas utile de chercher davantage sur xenocanto.org et apprécions votre coopération pour limiter la charge sur leurs serveurs.
- unlabeled_soundscapes/
Données audio non étiquetées provenant des mêmes lieux d'enregistrement que les paysages sonores de test.
- train_metadata.csv
Une large gamme de métadonnées est fournie pour les données d'entraînement.
- eBird_Taxonomy_v2021.csv
Données sur les relations entre différentes espèces.
Importation des modules¶
# Modules pour manipuler les fichiers audio
import librosa
import librosa.display
import matplotlib.pyplot as plt
import numpy as np
from IPython.display import Audio
# Geolocalisation
import plotly.express as px # peut-être pas
import geopandas as gpd
# Machine learning
from sklearn.cluster import KMeans # Et/ou un autre certainement
import pandas as pd
# Sauvegarde ou récupération de données
import json
import requests
# Divers affichages
from IPython.display import display, HTML
from wordcloud import WordCloud
import os
# Dénombrement
from collections import Counter
# Gestion des chaînes
from itertools import chain
import ast
import re
# Inclusion du géocodage inversé
from geopy.geocoders import Nominatim
import time
def summarize_dataframe(df):
# Vérification que les colonnes nécessaires sont présentes dans le DataFrame
if 'latitude' not in df.columns or 'longitude' not in df.columns:
raise ValueError("Les colonnes 'latitude' et 'longitude' sont requises dans le DataFrame.")
# Calcul du nombre total de lignes et de colonnes
num_rows, num_cols = df.shape
# Calcul du nombre de données manquantes pour les colonnes spécifiées
missing_counts = df[['latitude', 'longitude']].isnull().sum()
# Calcul du pourcentage de données manquantes pour les colonnes spécifiées
missing_percentage = (missing_counts / num_rows) * 100
# Création d'un DataFrame pour le résumé des colonnes spécifiques
summary_df = pd.DataFrame({
'Column Name': ['latitude', 'longitude'],
'Missing Data (%)': missing_percentage.round(2),
'Number': missing_counts
})
# Ajout des informations sur le nombre total de lignes et de colonnes au début du DataFrame
overall_info = pd.DataFrame({
'Column Name': ['Total Rows', 'Total Columns'],
'Missing Data (%)': ['-', '-'],
'Number': [num_rows, num_cols]
})
# Concaténation des informations générales avec le résumé des colonnes spécifiques
final_summary = pd.concat([overall_info, summary_df], ignore_index=True)
# Affichage du DataFrame sous forme de tableau HTML
display(HTML(final_summary.to_html(index=False)))
# Exemple d'utilisation
# summarize_dataframe(meta_data)
Chargement et sauvegarde de l'audio :
librosapermet de charger facilement des fichiers audio dans Python pour traitement, et de les sauvegarder après modification ou analyse.Extraction de caractéristiques : La bibliothèque fournit des outils pour extraire diverses caractéristiques audio telles que le spectre de puissance, la chroma (liée aux notes musicales), le mel-spectrogramme (qui est une représentation perceptuelle du spectre sonore), les coefficients cepstraux en fréquence de Mel (MFCCs), et plus encore.
Analyse du rythme :
librosapeut aider à détecter le tempo, les battements, et les temps forts dans un morceau de musique.Transformation et manipulation du son : Elle offre des fonctionnalités pour effectuer des transformations sur les signaux audio, telles que le stretching temporel (modification de la durée sans altérer la hauteur), le pitch shifting (modification de la hauteur sans changer la durée), et autres transformations.
Visualisation :
librosafournit des outils pour visualiser différentes représentations audio, ce qui peut aider dans l'analyse et la compréhension des données audio.Synthèse audio : Elle permet également la création de signaux audio par synthèse.
librosa est donc extrêmement utile pour ce qui nous intéresse ici, l'identification des espèces d'oiseaux à partir de leurs chants ou leurs cris.
Premier regard sur les fichiers audio¶
Spectogramme¶
Comment fonctionne un spectrogramme¶
Un spectrogramme est créé en divisant le signal audio en segments (fenêtrage) et en appliquant la transformée de Fourier sur chaque segment pour obtenir un ensemble de fréquences et leur amplitude respective. Ces segments sont ensuite visualisés les uns à côté des autres pour montrer comment le contenu fréquentiel change au fil du temps.
Les axes du spectrogramme représentent :
- L'axe horizontal (x-axis) : Il représente le temps.
- L'axe vertical (y-axis) : Il représente les fréquences.
- Les couleurs : Elles représentent l'amplitude ou l'intensité de chaque fréquence à un moment donné.
Types de spectrogrammes dans librosa¶
librosa offre différents types de spectrogrammes pour l'analyse du son :
Spectrogramme de puissance :
- C'est un spectrogramme classique où l'intensité de chaque fréquence est mesurée en puissance.
- Vous pouvez l'obtenir avec
librosa.stft().
Spectrogramme Mel :
- C'est un spectrogramme ajusté pour correspondre à la perception humaine de la hauteur sonore.
- Les fréquences sont mappées sur une échelle perceptuelle (Mel).
- Vous pouvez l'obtenir avec
librosa.feature.melspectrogram().
Spectrogramme de décibels :
- C'est un spectrogramme transformé en décibels pour correspondre davantage à la perception de l'intensité sonore par l'oreille humaine.
- Vous pouvez le convertir depuis un spectrogramme de puissance ou Mel en utilisant
librosa.power_to_db().
Utilisation pratique¶
librosa fournit plusieurs fonctions utiles pour manipuler et visualiser les spectrogrammes. Par exemple :
librosa.stft(): Calcule la transformée de Fourier sur des fenêtres du signal pour obtenir un spectrogramme de puissance.librosa.feature.melspectrogram(): Calcule un spectrogramme Mel.librosa.display.specshow(): Affiche le spectrogramme avec des options pour configurer les axes.librosa.power_to_db(): Convertit un spectrogramme en échelle logarithmique (décibels).
Ces outils sont largement utilisés dans le domaine de la musique, de l'audio et de la reconnaissance vocale pour analyser et comprendre les signaux sonores.
def load_audio(file_path):
audio, sr = librosa.load(file_path, sr=None)
return audio, sr
Dans le contexte du module librosa, sr est l'abréviation de "sample rate" ou "taux d'échantillonnage". Il s'agit du nombre d'échantillons audio par seconde dans le fichier audio chargé.
Le taux d'échantillonnage est important car il détermine la qualité audio et la fréquence maximale qui peut être représentée dans le fichier. Par exemple, un taux d'échantillonnage de 44 100 Hz (ou 44,1 kHz) est courant pour la musique et signifie que 44 100 échantillons sont enregistrés chaque seconde.
Lorsque vous chargez un fichier audio avec librosa.load(), vous pouvez spécifier le taux d'échantillonnage souhaité, mais par défaut, librosa le redimensionne à 22 050 Hz si vous ne spécifiez pas sr.
Ici, a priori, tous les fichiers audio auront un taux d'échantillonnage de 32 kHz.
def plot_spectrogram(filename):
# 1. Charger le fichier audio
y, sr = load_audio(filename)
# 2. Calculer le spectrogramme
S = librosa.feature.melspectrogram(y=y, sr=sr, n_mels=128)
# Convertir en échelle logarithmique
S_dB = librosa.power_to_db(S, ref=np.max)
# 3. Afficher le spectrogramme
plt.figure(figsize=(10, 4))
librosa.display.specshow(S_dB, sr=sr, x_axis='time', y_axis='mel')
plt.colorbar(format='%+2.0f dB')
plt.title('Spectrogramme mel')
plt.tight_layout()
plt.show()
Premier exemple simple¶
# Asian Brown Flycatcher : Gobemouche brun
bird1='train/asbfly/XC134896.ogg'
def audio_spectro(bird):
y, sr = load_audio(bird)
# Mettre en place un lecteur audio
audio = Audio(y, rate=sr)
plot_spectrogram(bird)
return audio
def audio(bird):
y, sr = load_audio(bird)
return y
audio_spectro(bird1)
L'appel ("call") du gobemouche brun a des motifs clairement identifié (6 ici)
L'échelle Mel est une échelle perceptuelle de hauteur sonore qui tente de correspondre à la perception humaine de la hauteur. Elle a été développée pour mieux représenter la façon dont les humains perçoivent les fréquences sonores, car notre perception de la hauteur sonore ne varie pas linéairement avec les fréquences.
Pourquoi l'échelle Mel est-elle utile ?¶
En acoustique et en traitement du signal, les fréquences plus élevées ne sont pas perçues comme étant proportionnellement plus aiguës, et il y a une diminution de la sensibilité de l'oreille humaine aux fréquences plus élevées. Pour refléter cela, l'échelle Mel compresse les fréquences plus élevées, de sorte qu'une variation d'une certaine quantité en haut de l'échelle est perçue de manière similaire à une variation plus grande en bas de l'échelle.
Comment l'échelle Mel est-elle utilisée ?¶
L'échelle Mel est fréquemment utilisée dans le traitement du signal audio, en particulier dans les applications de reconnaissance vocale et de musique, car elle reflète mieux notre perception de la hauteur sonore.
Spectrogrammes Mel : En utilisant un ensemble de filtres Mel, on peut transformer un spectrogramme classique en un spectrogramme Mel, qui est plus aligné sur la perception humaine.
MFCC (Cepstral Coefficients Mel-frequency) : C'est une transformation qui utilise l'échelle Mel pour extraire des caractéristiques des signaux audio, souvent utilisée dans la reconnaissance vocale et d'autres applications d'apprentissage automatique liées au son.
Conversion entre les fréquences et l'échelle Mel¶
La conversion entre les fréquences en Hertz et l'échelle Mel est effectuée avec des formules spécifiques. Une approximation courante est la suivante :
- Pour convertir des Hertz en Mel :
mel = 2595 * log10(1 + freq / 700) - Pour convertir des Mel en Hertz :
freq = 700 * (10^(mel / 2595) - 1)
Ces formules permettent de créer des filtres ou des échelles adaptés à la perception humaine, ce qui est souvent crucial dans le traitement audio.
Afficher une partie spécifique du spectrogramme¶
# Charger l'audio
y, sr = librosa.load(bird1)
# Calculer le spectrogramme
S = librosa.feature.melspectrogram(y=y, sr=sr, n_mels=128)
S_dB = librosa.power_to_db(S, ref=np.max)
# Définir les limites de temps pour l'affichage
start_time = 15 # en secondes
end_time = 16 # en secondes
start_frame = int(start_time * sr / 512) # Convertir les secondes en indices de frame
end_frame = int(end_time * sr / 512)
# Afficher le spectrogramme
plt.figure(figsize=(10, 4))
librosa.display.specshow(S_dB[:, start_frame:end_frame], sr=sr, x_axis='time', y_axis='mel')
plt.colorbar(format='%+2.0f dB')
plt.title('Spectrogramme mel')
plt.tight_layout()
plt.show()
Filtrer un intervalle de fréquence¶
# Utilisation de filtres passe-bande avec scipy
import scipy.signal
# Paramètres du filtre
low_freq = 7000 # Fréquence de coupure basse en Hz
high_freq = 11000 # Fréquence de coupure haute en Hz
# Créer un filtre passe-bande
sos = scipy.signal.butter(10, [low_freq, high_freq], btype='bandpass', fs=sr, output='sos')
# Appliquer le filtre
y_filtered = scipy.signal.sosfilt(sos, y)
# Calculer le spectrogramme
S = librosa.feature.melspectrogram(y=y_filtered, sr=sr, n_mels=128)
S_dB = librosa.power_to_db(S, ref=np.max)
# Afficher le spectrogramme en Hz
plt.figure(figsize=(10, 4))
librosa.display.specshow(S_dB, sr=sr, x_axis='time', y_axis='hz')
plt.colorbar(format='%+2.0f dB')
plt.title('Spectrogramme Mel filtré et affiché en Hz')
plt.tight_layout()
# Ajuster l'axe des fréquences
plt.ylim(low_freq, high_freq)
plt.show()
Filtrer un intervalle d'intensité en dB¶
# Calculer le spectrogramme
S = librosa.feature.melspectrogram(y=y, sr=sr, n_mels=128)
S_dB = librosa.power_to_db(S, ref=np.max)
# Définir les limites d'intensité en dB
min_dB = -20
max_dB = 0
# Créer un masque pour les valeurs en dehors de l'intervalle
mask = (S_dB >= min_dB) & (S_dB <= max_dB)
S_filtered = np.where(mask, S_dB, -80) # Remplacer les valeurs masquées par -80 dB (ou tout autre minimum souhaité)
# Afficher le spectrogramme
plt.figure(figsize=(10, 4))
librosa.display.specshow(S_filtered, sr=sr, x_axis='time', y_axis='mel')
plt.colorbar(format='%+2.0f dB')
plt.title('Spectrogramme mel filtré par intensité')
plt.tight_layout()
plt.show()
Second exemple avec des trilles¶
meta_data = pd.read_csv('birdclef-2024/train_metadata.csv')
# Oiseaux réputés pour leur trille
# Pinson des arbres
# meta_data[meta_data['common_name']=="Common Chaffinch"]
# Troglodyte mignon
# meta_data[meta_data['common_name']=="Eurasian Wren"]
# Rouge-Gorge
# meta_data[meta_data['common_name']=="European Robin"]
# Mésange charbonnière
# meta_data[meta_data['common_name']=="Great Tit"]
# Alouette des champs
# meta_data[meta_data['common_name']=='Skylark']
meta_data[meta_data['common_name']=='Yellow-billed Babbler']
| primary_label | secondary_labels | type | latitude | longitude | scientific_name | common_name | author | license | rating | url | filename | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 23901 | yebbab1 | [] | [''] | 6.3667 | 81.5167 | Argya affinis | Yellow-billed Babbler | Tero Linjama | Creative Commons Attribution-NonCommercial-Sha... | 5.0 | https://xeno-canto.org/164028 | yebbab1/XC164028.ogg |
| 23902 | yebbab1 | [] | [''] | 6.9944 | 80.4114 | Argya affinis | Yellow-billed Babbler | Andrew Spencer | Creative Commons Attribution-NonCommercial-Sha... | 5.0 | https://xeno-canto.org/190813 | yebbab1/XC190813.ogg |
| 23903 | yebbab1 | [] | [''] | NaN | NaN | Argya affinis | Yellow-billed Babbler | David Farrow | Creative Commons Attribution-NonCommercial-Sha... | 5.0 | https://xeno-canto.org/19927 | yebbab1/XC19927.ogg |
| 23904 | yebbab1 | [] | [''] | 6.3161 | 81.4830 | Argya affinis | Yellow-billed Babbler | Mike Nelson | Creative Commons Attribution-NonCommercial-Sha... | 4.0 | https://xeno-canto.org/207876 | yebbab1/XC207876.ogg |
| 23905 | yebbab1 | [] | [''] | 12.9912 | 80.2363 | Argya affinis | Yellow-billed Babbler | Vivek Puliyeri | Creative Commons Attribution-NonCommercial-Sha... | 4.0 | https://xeno-canto.org/267098 | yebbab1/XC267098.ogg |
| 23906 | yebbab1 | [] | [''] | 10.3437 | 77.2184 | Argya affinis | Yellow-billed Babbler | Dilip KG | Creative Commons Attribution-NonCommercial-Sha... | 3.0 | https://xeno-canto.org/284848 | yebbab1/XC284848.ogg |
| 23907 | yebbab1 | [] | [''] | 9.8484 | 78.1699 | Argya affinis | Yellow-billed Babbler | Vijay Nivash | Creative Commons Attribution-NonCommercial-Sha... | 5.0 | https://xeno-canto.org/318440 | yebbab1/XC318440.ogg |
| 23908 | yebbab1 | [] | [''] | 6.9966 | 80.4151 | Argya affinis | Yellow-billed Babbler | Jim Holmes | Creative Commons Attribution-NonCommercial-Sha... | 5.0 | https://xeno-canto.org/346136 | yebbab1/XC346136.ogg |
| 23909 | yebbab1 | [] | [''] | 6.9966 | 80.4151 | Argya affinis | Yellow-billed Babbler | Jim Holmes | Creative Commons Attribution-NonCommercial-Sha... | 5.0 | https://xeno-canto.org/346138 | yebbab1/XC346138.ogg |
| 23910 | yebbab1 | [] | [''] | 12.0014 | 79.8178 | Argya affinis | Yellow-billed Babbler | bernard Fort | Creative Commons Attribution-NonCommercial-Sha... | 4.0 | https://xeno-canto.org/392180 | yebbab1/XC392180.ogg |
| 23911 | yebbab1 | [] | [''] | 11.0920 | 76.7878 | Argya affinis | Yellow-billed Babbler | Viral joshi | Creative Commons Attribution-NonCommercial-Sha... | 5.0 | https://xeno-canto.org/399020 | yebbab1/XC399020.ogg |
| 23912 | yebbab1 | [] | [''] | 11.0477 | 76.8642 | Argya affinis | Yellow-billed Babbler | Dilip KG | Creative Commons Attribution-NonCommercial-Sha... | 4.0 | https://xeno-canto.org/406116 | yebbab1/XC406116.ogg |
| 23913 | yebbab1 | [] | [''] | 13.3470 | 74.7880 | Argya affinis | Yellow-billed Babbler | ARUN PRABHU | Creative Commons Attribution-NonCommercial-Sha... | 0.0 | https://xeno-canto.org/424699 | yebbab1/XC424699.ogg |
| 23914 | yebbab1 | [] | [''] | 12.8318 | 80.0382 | Argya affinis | Yellow-billed Babbler | Sreekumar Chirukandoth | Creative Commons Attribution-NonCommercial-Sha... | 4.5 | https://xeno-canto.org/444696 | yebbab1/XC444696.ogg |
| 23915 | yebbab1 | [] | [''] | 13.0206 | 77.5701 | Argya affinis | Yellow-billed Babbler | Isheta divya | Creative Commons Attribution-NonCommercial-Sha... | 2.5 | https://xeno-canto.org/465497 | yebbab1/XC465497.ogg |
| 23916 | yebbab1 | [] | [''] | 12.9912 | 80.2363 | Argya affinis | Yellow-billed Babbler | Vivek Puliyeri | Creative Commons Attribution-NonCommercial-Sha... | 4.0 | https://xeno-canto.org/469375 | yebbab1/XC469375.ogg |
| 23917 | yebbab1 | [] | [''] | 12.9912 | 80.2363 | Argya affinis | Yellow-billed Babbler | Vivek Puliyeri | Creative Commons Attribution-NonCommercial-Sha... | 4.0 | https://xeno-canto.org/469376 | yebbab1/XC469376.ogg |
| 23918 | yebbab1 | [] | [''] | 17.4054 | 78.5317 | Argya affinis | Yellow-billed Babbler | Karol Kustusch | Creative Commons Attribution-NonCommercial-Sha... | 4.0 | https://xeno-canto.org/531087 | yebbab1/XC531087.ogg |
| 23919 | yebbab1 | [] | [''] | 12.0264 | 79.8519 | Argya affinis | Yellow-billed Babbler | Romit Singha Roy | Creative Commons Attribution-NonCommercial-Sha... | 0.0 | https://xeno-canto.org/536074 | yebbab1/XC536074.ogg |
| 23920 | yebbab1 | [] | [''] | 12.1407 | 75.1703 | Argya affinis | Yellow-billed Babbler | Jayakrishnan U | Creative Commons Attribution-NonCommercial-Sha... | 5.0 | https://xeno-canto.org/545194 | yebbab1/XC545194.ogg |
| 23921 | yebbab1 | [] | [''] | 12.1407 | 75.1703 | Argya affinis | Yellow-billed Babbler | Jayakrishnan U | Creative Commons Attribution-NonCommercial-Sha... | 3.5 | https://xeno-canto.org/545205 | yebbab1/XC545205.ogg |
| 23922 | yebbab1 | [] | [''] | 6.8684 | 81.0458 | Argya affinis | Yellow-billed Babbler | Blair Jollands | Creative Commons Attribution-NonCommercial-Sha... | 5.0 | https://xeno-canto.org/588845 | yebbab1/XC588845.ogg |
| 23923 | yebbab1 | [] | [''] | 6.8684 | 81.0458 | Argya affinis | Yellow-billed Babbler | Blair Jollands | Creative Commons Attribution-NonCommercial-Sha... | 5.0 | https://xeno-canto.org/588850 | yebbab1/XC588850.ogg |
| 23924 | yebbab1 | ['revbul', 'rewbul'] | ['call'] | 11.5313 | 76.6474 | Argya affinis | Yellow-billed Babbler | Jelle Scharringa | Creative Commons Attribution-NonCommercial-Sha... | 4.5 | http://xeno-canto.org/670334 | yebbab1/XC670334.ogg |
| 23925 | yebbab1 | [] | ['adult', 'call', 'sex uncertain'] | 8.5831 | 81.2104 | Argya affinis | Yellow-billed Babbler | Vihansith Kulatunga | Creative Commons Attribution-ShareAlike 4.0 | 5.0 | http://xeno-canto.org/680927 | yebbab1/XC680927.ogg |
| 23926 | yebbab1 | [] | [''] | 10.3027 | 77.1805 | Argya affinis | Yellow-billed Babbler | Sathyan Meppayur | Creative Commons Attribution-NonCommercial-Sha... | 5.0 | https://xeno-canto.org/741609 | yebbab1/XC741609.ogg |
| 23927 | yebbab1 | [] | [''] | 6.9940 | 80.4113 | Argya affinis | Yellow-billed Babbler | Okamoto Keita Sin | Creative Commons Attribution-NonCommercial-Sha... | 5.0 | https://xeno-canto.org/756556 | yebbab1/XC756556.ogg |
| 23928 | yebbab1 | [] | [''] | 6.9696 | 80.7684 | Argya affinis | Yellow-billed Babbler | Okamoto Keita Sin | Creative Commons Attribution-NonCommercial-Sha... | 5.0 | https://xeno-canto.org/756557 | yebbab1/XC756557.ogg |
# Timalie à bec jaune (Yellow-billed Babbler, Turdoides affinis) :
# Ces oiseaux ont des trilles distinctives dans leur communication
bird2 = 'train/yebbab1/XC164028.ogg'
audio_spectro(bird2)
Les trilles du Timalie sont nombreuses, très rapprochées et diversifiées
Premier regard sur les données¶
train_metadata.csv¶
meta_data.head(4)
| primary_label | secondary_labels | type | latitude | longitude | scientific_name | common_name | author | license | rating | url | filename | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | asbfly | [] | ['call'] | 39.2297 | 118.1987 | Muscicapa dauurica | Asian Brown Flycatcher | Matt Slaymaker | Creative Commons Attribution-NonCommercial-Sha... | 5.0 | https://www.xeno-canto.org/134896 | asbfly/XC134896.ogg |
| 1 | asbfly | [] | ['song'] | 51.4030 | 104.6401 | Muscicapa dauurica | Asian Brown Flycatcher | Magnus Hellström | Creative Commons Attribution-NonCommercial-Sha... | 2.5 | https://www.xeno-canto.org/164848 | asbfly/XC164848.ogg |
| 2 | asbfly | [] | ['song'] | 36.3319 | 127.3555 | Muscicapa dauurica | Asian Brown Flycatcher | Stuart Fisher | Creative Commons Attribution-NonCommercial-Sha... | 2.5 | https://www.xeno-canto.org/175797 | asbfly/XC175797.ogg |
| 3 | asbfly | [] | ['call'] | 21.1697 | 70.6005 | Muscicapa dauurica | Asian Brown Flycatcher | vir joshi | Creative Commons Attribution-NonCommercial-Sha... | 4.0 | https://www.xeno-canto.org/207738 | asbfly/XC207738.ogg |
meta_data.info()
<class 'pandas.core.frame.DataFrame'> RangeIndex: 24459 entries, 0 to 24458 Data columns (total 12 columns): # Column Non-Null Count Dtype --- ------ -------------- ----- 0 primary_label 24459 non-null object 1 secondary_labels 24459 non-null object 2 type 24459 non-null object 3 latitude 24081 non-null float64 4 longitude 24081 non-null float64 5 scientific_name 24459 non-null object 6 common_name 24459 non-null object 7 author 24459 non-null object 8 license 24459 non-null object 9 rating 24459 non-null float64 10 url 24459 non-null object 11 filename 24459 non-null object dtypes: float64(3), object(9) memory usage: 2.2+ MB
Données manquantes¶
Quelques centaines d'enregistrements ne sont pas localisés. On va tenter de réaliser une imputation du pays concerné après avoir créé une colonne dédiée.
meta_data.latitude.isna().value_counts()
latitude False 24081 True 378 Name: count, dtype: int64
378 données géographiques manquantes
Nombre d'espèces d'oiseaux¶
len(meta_data.primary_label.unique())
182
# Vérification
print(len(meta_data.scientific_name.unique()))
print(len(meta_data.common_name.unique()))
182 182
meta_data.primary_label.value_counts()
primary_label
zitcis1 500
lirplo 500
litgre1 500
comgre 500
comkin1 500
...
blaeag1 6
wynlau1 6
niwpig1 5
asiope1 5
integr 5
Name: count, Length: 182, dtype: int64
Fiche enregistrement¶
# Fonction qui permet de récupérer l'altitude d'un lieu
def get_elevation(lat, lng):
try:
url = f"https://api.opentopodata.org/v1/eudem25m?locations={lat},{lng}"
response = requests.get(url)
data = response.json()
if data['status'] == 'OK':
elevation = data['results'][0]['elevation']
return elevation
except Exception as e:
print(f"Error retrieving data for location {lat}, {lng}: {e}")
return None
# Fonction qui permet de récupérer le pays d'un lieu
geolocator = Nominatim(user_agent="geoapiExercises")
def get_country(latitude, longitude):
"""Fonction auxiliaire pour obtenir le pays depuis des coordonnées GPS."""
try:
location = geolocator.reverse((latitude, longitude), exactly_one=True, language='en')
return location.raw['address']['country']
except:
return "Country not available"
# Fonction qui affiche une fiche de renseignements détaillés à propos d'un enregistrement sonore et de l'oiseau concerné
def retrieve_bird_details(common_name):
# Filtrer les données pour le nom commun spécifié
bird_data = meta_data[meta_data['common_name'] == common_name]
# Vérifier si des enregistrements existent pour cet oiseau
if bird_data.empty:
return f"Aucun enregistrement trouvé pour l'oiseau nommé '{common_name}'."
# Calculer le nombre total d'enregistrements
total_records = len(bird_data)
# Créer la fiche détaillée pour le premier enregistrement trouvé (exemple)
bird_record = bird_data.iloc[0]
# Nettoyer le type d'enregistrement
types = bird_record['type'].strip("[]").replace("'", "")
# Nettoyer et compter les types d'enregistrements
type_counts = bird_data['type'].apply(lambda x: x.strip("[]").replace("'", "").strip()).value_counts().to_dict()
# Préparer la section de comptage des types pour l'affichage
type_counts_str = ', '.join([f"{key}: {value}" for key, value in type_counts.items()])
# Obtenir les coordonnées
latitude = bird_record['latitude']
longitude = bird_record['longitude']
# Géocodage inversé pour obtenir le pays
country = get_country(latitude, longitude)
# Attendre un peu pour respecter les limites de taux du service de géocodage
time.sleep(1)
# Géocoder et compter les enregistrements par pays
'''country_counts = {}
for _, row in bird_data.iterrows():
country = get_country(row['latitude'], row['longitude'])
if country in country_counts:
country_counts[country] += 1
else:
country_counts[country] = 1
time.sleep(1) # Limiter la fréquence des requêtes pour respecter les politiques d'utilisation
country_counts_str = ', '.join([f"{key}: {value}" for key, value in country_counts.items()])'''
details_html = f"""
<p><strong>Nom commun:</strong> {bird_record['common_name']}</p>
<p><strong>Nom scientifique:</strong> {bird_record['scientific_name']}</p>
<p><strong>Type de son:</strong> {types}</p>
<p><strong>Coordonnées GPS:</strong> {latitude}, {longitude} (<strong>Pays:</strong> {country})</p>
<p><strong>Auteur:</strong> {bird_record['author']}</p>
<p><strong>URL de l'enregistrement:</strong> <a href='{bird_record['url']}'>{bird_record['url']}</a></p>
<p><strong>Qualité de l'enregistrement:</strong> {bird_record['rating']}</p>
<p><strong>Nombre total d'enregistrements pour '{common_name}':</strong> {total_records}</p>
<p><strong>Détail des types d'enregistrements:</strong> {type_counts_str}</p>
<p><strong>Fichier audio:</strong> <a href='birdclef-2024/train_audio/{bird_record['filename']}'>Écouter le premier enregistrement</a></p>
"""
return details_html
# Si besoin
# <p><strong>Enregistrements par pays:</strong> {country_counts_str}</p>
# Test de la fonction avec un exemple en HTML
display(HTML(retrieve_bird_details("Asian Brown Flycatcher")))
Nom commun: Asian Brown Flycatcher
Nom scientifique: Muscicapa dauurica
Type de son: call
Coordonnées GPS: 39.2297, 118.1987 (Pays: China)
Auteur: Matt Slaymaker
URL de l'enregistrement: https://www.xeno-canto.org/134896
Qualité de l'enregistrement: 5.0
Nombre total d'enregistrements pour 'Asian Brown Flycatcher': 105
Détail des types d'enregistrements: : 44, call: 26, song: 14, Call: 5, adult, call, sex uncertain: 4, call, male, song: 2, alarm call: 2, clicks its bill: 1, male, song: 1, adult, call: 1, Song: 1, Subsong: 1, Alarm call: 1, adult, call, sex uncertain, song: 1, call, life stage uncertain, sex uncertain: 1
Fichier audio: Écouter le premier enregistrement
meta_data.describe()
| latitude | longitude | rating | |
|---|---|---|---|
| count | 24081.000000 | 24081.000000 | 24459.000000 |
| mean | 32.537040 | 43.640699 | 3.843493 |
| std | 19.440382 | 50.191352 | 1.100840 |
| min | -43.524000 | -171.765400 | 0.000000 |
| 25% | 17.160100 | 2.545700 | 3.000000 |
| 50% | 37.155100 | 26.687600 | 4.000000 |
| 75% | 49.114400 | 85.319300 | 5.000000 |
| max | 71.964000 | 177.447800 | 5.000000 |
L'essentiel de la planète est couvert.
Pour la qualité (rating) des enregistrements on y reviendra.
eBird_Taxonomy_v2021.csv¶
taxonomy = pd.read_csv('birdclef-2024/eBird_Taxonomy_v2021.csv')
taxonomy.head(4)
| TAXON_ORDER | CATEGORY | SPECIES_CODE | PRIMARY_COM_NAME | SCI_NAME | ORDER1 | FAMILY | SPECIES_GROUP | REPORT_AS | |
|---|---|---|---|---|---|---|---|---|---|
| 0 | 1 | species | ostric2 | Common Ostrich | Struthio camelus | Struthioniformes | Struthionidae (Ostriches) | Ostriches | NaN |
| 1 | 6 | species | ostric3 | Somali Ostrich | Struthio molybdophanes | Struthioniformes | Struthionidae (Ostriches) | NaN | NaN |
| 2 | 7 | slash | y00934 | Common/Somali Ostrich | Struthio camelus/molybdophanes | Struthioniformes | Struthionidae (Ostriches) | NaN | NaN |
| 3 | 8 | species | grerhe1 | Greater Rhea | Rhea americana | Rheiformes | Rheidae (Rheas) | Rheas | NaN |
taxonomy.info()
<class 'pandas.core.frame.DataFrame'> RangeIndex: 16753 entries, 0 to 16752 Data columns (total 9 columns): # Column Non-Null Count Dtype --- ------ -------------- ----- 0 TAXON_ORDER 16753 non-null int64 1 CATEGORY 16753 non-null object 2 SPECIES_CODE 16753 non-null object 3 PRIMARY_COM_NAME 16753 non-null object 4 SCI_NAME 16753 non-null object 5 ORDER1 16751 non-null object 6 FAMILY 16740 non-null object 7 SPECIES_GROUP 216 non-null object 8 REPORT_AS 3876 non-null object dtypes: int64(1), object(8) memory usage: 1.2+ MB
taxonomy.SPECIES_GROUP.value_counts()
SPECIES_GROUP
Ostriches 1
African Warblers 1
Australasian Robins 1
Rockfowl 1
Rockjumpers 1
..
Asities 1
Old World Pittas 1
Antbirds 1
Crescentchests 1
Others 1
Name: count, Length: 216, dtype: int64
Après observation rapide cette colonne met au pluriel le nom central de PRIMARY_COM_NAME (Ostrich --> Ostriches etc). L'intérêt semble nul donc on va supprimer cette colonne.
taxonomy.REPORT_AS.unique()
array([nan, 'lesrhe2', 'higtin1', ..., 'banana', 'cubbul1', 'woofin1'],
dtype=object)
Report as = signalé comme
Par exemple zitcis2 est signalé comme zitcis1 et zebfin1 comme zebfin2. La pertinence de conserver cette colonne restant à démontrer on va la supprimer.
Suppression des 2 colonnes¶
taxonomy = taxonomy.drop(['SPECIES_GROUP', 'REPORT_AS'], axis=1)
Fusion des 2 dataframes¶
# Fusion des dataframes sur les colonnes spécifiées
birds_df = pd.merge(taxonomy, meta_data, left_on='SCI_NAME', right_on='scientific_name', how='inner')
# Affichage des premières lignes du dataframe fusionné pour vérifier le résultat
birds_df.head()
| TAXON_ORDER | CATEGORY | SPECIES_CODE | PRIMARY_COM_NAME | SCI_NAME | ORDER1 | FAMILY | primary_label | secondary_labels | type | latitude | longitude | scientific_name | common_name | author | license | rating | url | filename | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 233 | species | lewduc1 | Lesser Whistling-Duck | Dendrocygna javanica | Anseriformes | Anatidae (Ducks, Geese, and Waterfowl) | lewduc1 | [] | ['call'] | 13.9144 | 104.5538 | Dendrocygna javanica | Lesser Whistling-Duck | Patrik Åberg | Creative Commons Attribution-NonCommercial-Sha... | 5.0 | https://www.xeno-canto.org/124374 | lewduc1/XC124374.ogg |
| 1 | 233 | species | lewduc1 | Lesser Whistling-Duck | Dendrocygna javanica | Anseriformes | Anatidae (Ducks, Geese, and Waterfowl) | lewduc1 | [] | ['flight call', 'song'] | 20.2609 | 100.0315 | Dendrocygna javanica | Lesser Whistling-Duck | Greg Irving | Creative Commons Attribution-NonCommercial-Sha... | 4.0 | https://www.xeno-canto.org/169762 | lewduc1/XC169762.ogg |
| 2 | 233 | species | lewduc1 | Lesser Whistling-Duck | Dendrocygna javanica | Anseriformes | Anatidae (Ducks, Geese, and Waterfowl) | lewduc1 | [] | ['call'] | 6.2917 | 81.2685 | Dendrocygna javanica | Lesser Whistling-Duck | Andrew Spencer | Creative Commons Attribution-NonCommercial-Sha... | 5.0 | https://www.xeno-canto.org/190865 | lewduc1/XC190865.ogg |
| 3 | 233 | species | lewduc1 | Lesser Whistling-Duck | Dendrocygna javanica | Anseriformes | Anatidae (Ducks, Geese, and Waterfowl) | lewduc1 | [] | ['call'] | 6.2917 | 81.2685 | Dendrocygna javanica | Lesser Whistling-Duck | Andrew Spencer | Creative Commons Attribution-NonCommercial-Sha... | 5.0 | https://www.xeno-canto.org/190866 | lewduc1/XC190866.ogg |
| 4 | 233 | species | lewduc1 | Lesser Whistling-Duck | Dendrocygna javanica | Anseriformes | Anatidae (Ducks, Geese, and Waterfowl) | lewduc1 | [] | ['call'] | 6.1702 | 81.2054 | Dendrocygna javanica | Lesser Whistling-Duck | Andrew Spencer | Creative Commons Attribution-NonCommercial-Sha... | 5.0 | https://www.xeno-canto.org/190973 | lewduc1/XC190973.ogg |
birds_df.info()
<class 'pandas.core.frame.DataFrame'> RangeIndex: 24459 entries, 0 to 24458 Data columns (total 19 columns): # Column Non-Null Count Dtype --- ------ -------------- ----- 0 TAXON_ORDER 24459 non-null int64 1 CATEGORY 24459 non-null object 2 SPECIES_CODE 24459 non-null object 3 PRIMARY_COM_NAME 24459 non-null object 4 SCI_NAME 24459 non-null object 5 ORDER1 24459 non-null object 6 FAMILY 24459 non-null object 7 primary_label 24459 non-null object 8 secondary_labels 24459 non-null object 9 type 24459 non-null object 10 latitude 24081 non-null float64 11 longitude 24081 non-null float64 12 scientific_name 24459 non-null object 13 common_name 24459 non-null object 14 author 24459 non-null object 15 license 24459 non-null object 16 rating 24459 non-null float64 17 url 24459 non-null object 18 filename 24459 non-null object dtypes: float64(3), int64(1), object(15) memory usage: 3.5+ MB
Aucune perte de données
birds_df.describe()
| TAXON_ORDER | latitude | longitude | rating | |
|---|---|---|---|---|
| count | 24459.000000 | 24081.000000 | 24081.000000 | 24459.000000 |
| mean | 14155.552435 | 32.537040 | 43.640699 | 3.843493 |
| std | 9718.624339 | 19.440382 | 50.191352 | 1.100840 |
| min | 233.000000 | -43.524000 | -171.765400 | 0.000000 |
| 25% | 6028.000000 | 17.160100 | 2.545700 | 3.000000 |
| 50% | 9389.000000 | 37.155100 | 26.687600 | 4.000000 |
| 75% | 23291.000000 | 49.114400 | 85.319300 | 5.000000 |
| max | 31096.000000 | 71.964000 | 177.447800 | 5.000000 |
# Point d'étape
summarize_dataframe(birds_df)
| Column Name | Missing Data (%) | Number |
|---|---|---|
| Total Rows | - | 24459 |
| Total Columns | - | 19 |
| latitude | 1.55 | 378 |
| longitude | 1.55 | 378 |
Regard plus poussé sur les données¶
- train_metadata.csv : Une large gamme de métadonnées est fournie pour les données d'entraînement. Les champs les plus directement pertinents sont :
-- primary_label : un code pour l'espèce d'oiseau. Vous pouvez consulter des informations détaillées sur les codes d'oiseaux en ajoutant le code à https://ebird.org/species/, comme https://ebird.org/species/amecro pour le Corbeau d'Amérique. Toutes les espèces n'ont pas leurs propres pages ; certains liens peuvent ne pas fonctionner.
-- latitude & longitude : coordonnées de l'endroit où l'enregistrement a été pris. Certaines espèces d'oiseaux peuvent avoir des 'dialectes' locaux d'appels, donc vous pourriez vouloir chercher une diversité géographique dans vos données d'entraînement.
-- auteur : L'utilisateur qui a fourni l'enregistrement.
-- filename : le nom du fichier audio associé.
- eBird_Taxonomy_v2021.csv - Données sur les relations entre différentes espèces.
Data Description:
Data Description for Train Metadata
| Feature Name | Data Type | Description |
|---|---|---|
| primary_label | Object | Primary label associated with the bird species |
| secondary_labels | Object | Secondary labels associated with the bird species |
| type | Object | Type or category of the recording(song, call etc) |
| latitude | Float64 | Latitude coordinates of recording location |
| longitude | Float64 | Longitude coordinates of recording location |
| scientific_name | Object | Scientific name of the bird species |
| common_name | Object | Common name of the bird species |
| author | Object | User who provided the recording |
| license | Object | Licensing terms associated with the recording |
| rating | Float64 | Rating assigned to the recording |
| url | Object | URL associated with the recording |
| filename | Object | Filename of the audio file |
Data Description for Bird Taxonomy
| Feature Name | Data Type | Description |
|---|---|---|
| TAXON_ORDER | Int64 | Taxonomic order # of the bird species |
| CATEGORY | Object | Category of the bird species |
| SPECIES_CODE | Object | Unique code assigned to the bird species |
| PRIMARY_COM_NAME | Object | Primary common name of the bird species |
| SCI_NAME | Object | Scientific name of the bird species |
| ORDER1 | Object | Taxonomic order of the bird species |
| FAMILY | Object | Taxonomic family of the bird species |
| SPECIES_GROUP | Object | Species group or category |
| REPORT_AS | Object | Additional information about the bird species |
Données geospatiales manquantes¶
# Calculer le nombre de valeurs manquantes pour les colonnes latitude et longitude par auteur
missing_data_by_author = birds_df[birds_df['latitude'].isna() | birds_df['longitude'].isna()].groupby('author').size()
# Trier les auteurs par le nombre de données manquantes en ordre décroissant
missing_data_by_author_sorted = missing_data_by_author.sort_values(ascending=False)
missing_data_by_author_sorted
author
José Carlos Sires 41
brendan sloan 31
Sathyan Meppayur 21
David Farrow 19
KALYAN SINGH SAJWAN 16
..
Jerome Fischer 1
Jennifer Luk 1
Jan Erik Kikkert 1
Jan Andersson 1
王自堃 1
Length: 104, dtype: int64
Est-ce que les 5 auteurs ci-dessus ont effectué d'autres enregistrements ?
# Liste des auteurs à analyser
top_authors = ["José Carlos Sires", "brendan sloan", "Sathyan Meppayur", "David Farrow", "KALYAN SINGH SAJWAN"]
# Calcul du nombre total d'enregistrements pour chaque auteur
total_records_by_top_authors = birds_df[birds_df['author'].isin(top_authors)].groupby('author').size().reset_index(name='Total Records')
# Calcul du nombre d'enregistrements sans données GPS pour chaque auteur
missing_gps_by_top_authors = birds_df[(birds_df['author'].isin(top_authors)) & (birds_df['latitude'].isna() | birds_df['longitude'].isna())]
missing_gps_counts = missing_gps_by_top_authors.groupby('author').size().reset_index(name='Missing GPS Records')
# Fusionner les deux DataFrames sur l'auteur
author_records_df = pd.merge(total_records_by_top_authors, missing_gps_counts, on='author', how='left')
# Remplir les valeurs manquantes avec 0 pour les auteurs sans enregistrements manquants de GPS
author_records_df['Missing GPS Records'] = author_records_df['Missing GPS Records'].fillna(0)
# Calcul du pourcentage de données GPS manquantes pour chaque auteur
author_records_df['Percentage Missing GPS'] = (author_records_df['Missing GPS Records'] / author_records_df['Total Records']) * 100
author_records_df
| author | Total Records | Missing GPS Records | Percentage Missing GPS | |
|---|---|---|---|---|
| 0 | David Farrow | 104 | 19 | 18.269231 |
| 1 | José Carlos Sires | 915 | 41 | 4.480874 |
| 2 | KALYAN SINGH SAJWAN | 18 | 16 | 88.888889 |
| 3 | Sathyan Meppayur | 102 | 21 | 20.588235 |
| 4 | brendan sloan | 31 | 31 | 100.000000 |
Dans quelle zones géographiques ces 5 (enfin 4) auteurs ont-ils effectués leurs enregistrements ? (Aucun espoir d'imputer les données manquantes de brenda sloan puisqu'aucun de ses enregistrements n'a de données GPS.)
# Extraire les données de latitude et longitude pour les auteurs sélectionnés
geo_data_by_author = birds_df[birds_df['author'].isin(top_authors)][['author', 'latitude', 'longitude']]
# Supprimer les entrées où la latitude ou la longitude sont manquantes pour une analyse précise des zones géographiques
geo_data_by_author = geo_data_by_author.dropna(subset=['latitude', 'longitude'])
# Calculer les plages de latitude et longitude pour chaque auteur
geo_range_by_author = geo_data_by_author.groupby('author').agg({
'latitude': ['min', 'max'],
'longitude': ['min', 'max']
})
geo_range_by_author
| latitude | longitude | |||
|---|---|---|---|---|
| min | max | min | max | |
| author | ||||
| David Farrow | 0.5606 | 57.39767 | -5.502434 | 123.6800 |
| José Carlos Sires | 32.7201 | 42.99520 | -9.000500 | -1.2221 |
| KALYAN SINGH SAJWAN | 29.2779 | 29.75240 | 78.526900 | 79.3399 |
| Sathyan Meppayur | 9.5087 | 15.46450 | 74.187900 | 77.2112 |
Les quatre auteurs ont effectué des enregistrements dans diverses zones géographiques, principalement situées selon les plages de latitude et de longitude suivantes :
- David Farrow :
Latitude : de 0.5606 à 57.39767 (couvrant des zones proches de l'équateur jusqu'au nord de l'Europe)
Longitude : de -5.502434 à 123.6800 (de l'ouest de l'Europe à l'est de l'Asie)
- José Carlos Sires :
Latitude : de 32.7201 à 42.99520 (principalement en Espagne et en Europe méridionale)
Longitude : de -9.0005 à -1.2221 (principalement en Espagne)
- KALYAN SINGH SAJWAN :
Latitude : de 29.2779 à 29.75240 (dans une région très spécifique du nord de l'Inde)
Longitude : de 78.526900 à 79.3399 (dans une région très spécifique du nord de l'Inde)
- Sathyan Meppayur :
Latitude : de 9.5087 à 15.4645 (principalement dans le sud de l'Inde)
Longitude : de 74.1879 à 77.2112 (principalement dans le sud de l'Inde)
- Il semble que brendan sloan n'ait pas de données valides de latitude et longitude enregistrées, car toutes ses données sont manquantes pour ces attributs.
# Sélection des auteurs pour lesquels nous allons imputer les données
authors_for_imputation = ["José Carlos Sires", "KALYAN SINGH SAJWAN", "Sathyan Meppayur"]
# Filtrer les données pour ces auteurs
data_to_impute = birds_df[birds_df['author'].isin(authors_for_imputation)]
# Calculer les moyennes des latitudes et longitudes pour ces auteurs
mean_coordinates = data_to_impute.groupby('author')[['latitude', 'longitude']].mean()
# Afficher les moyennes calculées pour vérification
mean_coordinates
| latitude | longitude | |
|---|---|---|
| author | ||
| José Carlos Sires | 37.131566 | -6.141302 |
| KALYAN SINGH SAJWAN | 29.515150 | 78.933400 |
| Sathyan Meppayur | 11.102235 | 76.301647 |
# Fonction pour appliquer l'imputation en utilisant les moyennes calculées
def impute_coordinates(row, mean_coords):
if pd.isna(row['latitude']):
row['latitude'] = mean_coords.loc[row['author'], 'latitude']
if pd.isna(row['longitude']):
row['longitude'] = mean_coords.loc[row['author'], 'longitude']
return row
# Appliquer l'imputation aux enregistrements sélectionnés
data_imputed = data_to_impute.apply(lambda row: impute_coordinates(row, mean_coordinates), axis=1)
# Vérifier si les valeurs ont été correctement imputées
data_imputed[(data_imputed['author'].isin(authors_for_imputation)) & (data_imputed['latitude'].isna() | data_imputed['longitude'].isna())]
| TAXON_ORDER | CATEGORY | SPECIES_CODE | PRIMARY_COM_NAME | SCI_NAME | ORDER1 | FAMILY | primary_label | secondary_labels | type | latitude | longitude | scientific_name | common_name | author | license | rating | url | filename |
|---|
# Réintégrer data_imputed dans birds_df en utilisant l'indexation avec .loc
birds_df.loc[data_imputed.index] = data_imputed
data_imputed.info()
<class 'pandas.core.frame.DataFrame'> Index: 1035 entries, 413 to 23471 Data columns (total 19 columns): # Column Non-Null Count Dtype --- ------ -------------- ----- 0 TAXON_ORDER 1035 non-null int64 1 CATEGORY 1035 non-null object 2 SPECIES_CODE 1035 non-null object 3 PRIMARY_COM_NAME 1035 non-null object 4 SCI_NAME 1035 non-null object 5 ORDER1 1035 non-null object 6 FAMILY 1035 non-null object 7 primary_label 1035 non-null object 8 secondary_labels 1035 non-null object 9 type 1035 non-null object 10 latitude 1035 non-null float64 11 longitude 1035 non-null float64 12 scientific_name 1035 non-null object 13 common_name 1035 non-null object 14 author 1035 non-null object 15 license 1035 non-null object 16 rating 1035 non-null float64 17 url 1035 non-null object 18 filename 1035 non-null object dtypes: float64(3), int64(1), object(15) memory usage: 161.7+ KB
# Point d'étape
summarize_dataframe(birds_df)
| Column Name | Missing Data (%) | Number |
|---|---|---|
| Total Rows | - | 24459 |
| Total Columns | - | 19 |
| latitude | 1.23 | 300 |
| longitude | 1.23 | 300 |
On pourrait traiter plus d'auteurs à données GPS manquantes mais l'effort ne semble pas indispensable d'autant que ces coordonnées ne sont pas accessibles dans les meta données des enregistrements tests. Il nous restera donc 300 enregistrements sans données géospatiales.
La colonne type¶
# Assurer que chaque élément de la colonne 'type' est une liste
birds_df['type'] = birds_df['type'].apply(lambda x: ast.literal_eval(x) if isinstance(x, str) else x)
# Aplatir la liste des listes en une seule liste
all_items = list(chain.from_iterable(birds_df['type']))
# Convertir en ensemble pour éliminer les doublons
unique_items = set(all_items)
# Si vous avez besoin d'une liste, vous pouvez convertir l'ensemble en liste
unique_items_list = list(unique_items)
print(unique_items_list)
['', 'colony song at mid afternoon', 'splash', 'Prrrr call', 'Strange sound', 'long trill', 'Voices', 'barks', 'rain', 'Interaction call', 'ti-ti-ti call', 'flight calls of a flock', 'calls during copulation?', 'calls at nest', 'breeding colony', 'house crow', 'hundreds of birds roosting', 'Fulvetta call?', 'nasal song', 'pi-peep', 'flight song', 'calls by pair', 'Male and Female in tandem', 'various types of calls', 'intraspecific agressive call', 'screeches', 'anthropogenic', 'calls', 'snarls', 'conflict song in flight', 'Some noises in the hide', 'Prrrrr call', 'song?', 'first time recorded in KSNP', 'call with breeding display of vertical flight and parachuting on a branch', 'roosting call', 'alarm call from a juvenile perched on a reed', 'wail', 'Begging calls from 3 nestlings in nest.', 'two different calls', 'sound of a colony', 'Call near the nest', 'c-c-c- call on 7-8 KHz', 'chamado de contato do bando', 'clucks', 'Circus aeruginosus imitations from a Eurasian Jay ?', 'Wing flaps', 'Mimicry call', 'aggressive flight call', 'antiphony song', 'Adults feeding babies', 'flighing along the creek', 'subsong', 'wing flaps', 'escape', 'like Purple Heron', 'sneezing', 'male call', 'caw', 'dive', 'calls at roost', 'in flight', 'at 1 and 10 sec.', "'predator scolding'", 'bill clappering', 'Cattle', 'ttt call', 'thin call', 'RED VENTED', 'Socializing in a group', 'human voice', 'Vocalization in pair formation.', 'advertising calls', 'sex uncertain', 'nest call', 'aggressive call', 'full song', 'imitations: Motacilla alba and Sylvia communis', 'Part of the call is similar to Layards Parakeet assuming that it mimics', 'call,flight call,begging call', 'Onomatopoeiac song', 'Matting', 'series of calls', 'nocturnal song', 'Alarm Call', 'noctural', 'noise of wings', 'aberrant song', 'a bit vibrated call', 'Call?', 'quite raspy call', 'fight of 2 males', '-night migration', 'begging call?', 'Droplets falling on tin roofs', 'no', 'Power station auxiliaries', 'communicating while feeding on the ground', 'conversational chatter', 'bird in hand', 'bisyllabic call', 'aberrant', 'hatchling or nestling', 'NFC', 'life stage uncertain', 'dry calls', 'Baby in nest', 'chatter calls', 'migration', 'long call', 'Wingbeats', 'winsong', 'wing flapping', 'Soft calls', 'Call when they were fighting', 'Adults and youngs', 'Wing noise', 'calls of a flushed bird', 'nocturnal migration', 'Near its nesting hole.', 'fledgling calls', 'including "ortolan-like" calls', 'zoo collection', 'group of several dozens roosting at dusk', '"tek" call', 'Alarm call', 'call from wire', 'Anthropogenic noise', 'separados por comas', 'take-off call', 'on 6-7 sec.', 'wing beat; cooing', 'copulation calls', 'mimicry', 'alarm call', 'courtship flight', '-', 'autumn call', 'Tyu-tyu-tyu calls of an alarmed flock', 'Call', 'Beats wings', 'adults and juveniles', 'Human noise', 'chicks begging', 'probably flight call', 'Mimicking call', 'Two wheeler', 'Maybe a call', 'anthropogenic:: rain', 'rolled calls', 'flock', 'female', 'Singing / Calling?', 'odd call', 'Brooding call', 'Common hawk cuckoo', 'Passing car', 'wing beat', 'only plik calls in migration', 'short call', 'My whistle to start the bird', 'desending series', 'dormitory', 'feeding call of juveniles', 'insects', 'Knock', 'Nest defending calls', 'Beat wings', 'Black eagle soaring in this area last three days.', 'gurgling', 'dawn song', 'anxiety call', 'flight', 'Dueting', 'human', 'calls on nest', 'Aggression', 'guttoral call', 'nocturnal flightcall', 'flight call', 'shout', 'contact call', 'Nocturnal roosting site', 'splashing', 'wingsong', 'screech', 'wings clapping', 'female calling youngs', 'non-contextual call', 'Boys', 'trekroep', 'aggressive calls', 'Roosting place', 'similar species sound can be hear f', 'Think it is a song', 'Investigating', 'wing-flaps', 'colony song at dusk', 'cricket', 'various calls', 'aggression', 'Courtship call/song', 'OBS NEW VOCALIZATION: CALL', 'Song?', 'clicks its bill', 'call or song?', 'creaky scream', 'Song', 'Youngs calls', 'Mimic', 'nocturnal', 'Duet', 'night; trekroep', 'thin social calls', 'flapping wings', 'wind', 'Territorial call', 'Girls', 'vehical.', 'quiet-call', 'mimicking a ChangeableHawkEagle', 'Flight call', 'male', 'Some strange sound', 'wing beats', 'Courtship calls', 'Nocturnal rooting site', 'rolled call', 'displaying', 'duet', 'agonistic call', 'social call', 'imitation tits (Parus spp) song', 'declare territory', 'flight calls of juveniles', 'duet calls', 'male displaying', 'group in flight', 'rock bush', 'juvenile call', 'wings claps', 'wing beats towards the end', 'song in flight', 'deviant night call', 'male and female calls', 'beack claping', 'call from the ground', 'I think it is a song', 'Lista de los tipos de sonidos', 'breeding birds', 'Purple Heron-like call', 'Mimicking Crested Hawk Eagle', 'Appeared to be courtship call', 'Prrrrrr call', 'anthropogenic:', 'not sure which kind of call', 'song,counter song', 'exciting call', 'goats', 'conflict call', 'Roost', 'sound', 'song', 'aerial display', 'nocturnal call', 'Human sound', 'Anthropogenic background noise', 'teaching nestling how we call', 'alarm call given to alert chicks', 'nocturnal flight call', 'No other sound', 'laughing', 'nocturnal flight call', '( First time seen Koyal Juvenile Bird with Pair of Crows)', 'wingbeats', 'pew call', 'short call in flight', 'hooting sound', 'alarm call? seemed to be close to nest.', 'street traffic', 'voice in conflict', 'Subsong', 'vehicle', 'investigative call', 'wing noise', 'metallic alarm', 'wingflaps', 'two differnt calls', 'night call', '?', 'Begging Call from a chick', 'water splashing', 'hoarse call', 'Fighting', 'agitated call', 'General Call', 'Adult call', 'rattle', 'croaks', 'Drumming', 'Song-B', 'weak call', 'growls', 'wings', 'high whistle', 'tt call', 'Possibly', 'secondary song', 'wing training', 'electric water motor pump', 'take-off and flight calls', 'trill', 'two types of calls', 'two callig types', 'chicks and adult in breeding colony', 'Calls from a flock', 'Nocturnal', 'alarm near young', 'bird chased by a Merlin', 'tttt call', 'Flight song', 'quiet conversations in a large flock on the bank', 'Male-female', 'two birds', '"M" call', 'two-note call', 'probably breeding call', 'alarm clapping with wings', 'short metallic call in flight', 'various N calls', 'it starts to fly', 'fighting', 'display flight', 'Rattle call', 'sleeping-place', 'an adult flying over it', "Birdwatcher's talking:mobiles ringing", 'wing flutter', 'juvenile', 'Conflict call', 'black-headed gull like call', 'begging call,juvenile', 'wing-beats', 'Nocturnal call', 'wingflapping', 'Thermal Power Station in service', 'Wingbeat', 'low dry calls', 'anxiety call (actor sparrowhawk)', 'raspy call', 'hunting', 'night', 'uncertain', 'low call', 'whistle', 'cooing', 'wings beats', 'pecking sound', 'Fight call', 'Song fro', 'quarrel call', 'takking call', 'voices', 'long whistles', 'metallic call', 'quiet song', 'sip', 'pii-pii-pii', 'Territorial calls.', 'rattling call', 'wins', 'high pitch', 'aggression call', 'autumn song', 'various calls - all listed in accompanying comments', 'advertising call', 'nocturnal migrant', 'Nocturnal flight call.', 'call', 'song notes while in flight', 'various call', 'very short variant', 'all of a sudden frightened', 'Call from a eucaliptus', '+ call juvenile', '"Mau"', 'call,flight call,alarm call,aggressive call', 'Displaying', 'single-note call', 'sound of bill closing', '"chi" call', '"kup" call', 'aggresive calls', 'Nocturnal flight call', 'Long Caw', '"tek" calls', 'voice of nestlings', 'alarm', 'chatter call', 'chicks calling', 'agressive', 'wingbeat', 'both the single and rapid long call', 'diving', 'rattling', 'buzzy juv/fledgling', '"tew" calls', 'Grraw', 'song,call', '"Todoit"', 'adult', 'Roost calls', 'Caw', 'Cattle in water', 'Bird in migration', 'watersplash', 'threat call', '"migrating bird"', 'wing flapping display', 'Wing whir', 'sounds from flapping wings', 'Taj Mahal:', 'mating call', 'breeding call', 'drumming', 'given in flight', 'nasal call', 'mimicry/imitation', 'chorus of a flock', 'Insect sounds in background', 'hostile', 'boat noise', 'subadult', 'migrating bird', 'chick', 'interaction calls', 'wings flapping sound', 'night flight call', 'human voices', 'wing hits', 'soft call', 'traffic', 'excitement call', 'short calls', 'calls?', '"tsiuu" call', 'comparison between both species migrating together', 'différents types of flight call', 'begging call', 'group song', 'Prrru call', 'roaring calls', 'bickering', 'Male', 'Mating Call', 'Nocturnal Flight Call', 'calls of hatchlings', 'flight calls', 'Misleading rolled calls', "bird's call with some grasscracking sound in the bird's voice.", 'investigating', 'taking off', 'trills', 'Interspersed talks of people for few seconds', 'chorus', 'group', 'migration call', 'whistles', 'lapwing-like call', 'Single Note Calls', 'Wing flapping', 'imitation Carduelis carduelis calls', '"ti-di"', 'Anthropogenic']
# Fonction pour nettoyer les mots en retirant la ponctuation
def clean_word(word):
return re.sub(r'[^\w\s]', '', word.lower())
# Aplatir la liste en assumant qu'elle peut contenir des sous-listes imbriquées
flat_list = ' '.join(all_items)
# Nettoyer chaque mot: supprimer les espaces superflus et les caractères spéciaux
cleaned_words = [clean_word(word) for word in all_items if word.strip()]
# Créer une chaîne de tous les mots uniques
words_string = ' '.join(cleaned_words)
# Créer l'objet WordCloud
wordcloud = WordCloud(width=800, height=400, background_color='white').generate(words_string)
# Afficher le nuage de mots généré
plt.figure(figsize=(10, 5))
plt.imshow(wordcloud, interpolation='bilinear')
plt.axis("off")
plt.show()
# Compter la fréquence de chaque mot dans l'ensemble des données
word_counts = Counter(word for word in cleaned_words)
# Convertir le compteur en DataFrame et trier par fréquence décroissante
word_counts_df = pd.DataFrame(word_counts.items(), columns=['Word', 'Frequency']).sort_values(by='Frequency', ascending=False).reset_index(drop=True)
# Filtrer le DataFrame pour exclure les lignes où le mot est un espace vide ou nul
word_counts_df_clean = word_counts_df[word_counts_df['Word'].str.strip() != '']
# Afficher le DataFrame nettoyé
word_counts_df_clean.reset_index(drop=True, inplace=True)
word_counts_df_clean
| Word | Frequency | |
|---|---|---|
| 0 | call | 8948 |
| 1 | song | 5844 |
| 2 | flight call | 3463 |
| 3 | male | 1919 |
| 4 | nocturnal flight call | 1613 |
| ... | ... | ... |
| 428 | beack claping | 1 |
| 429 | dive | 1 |
| 430 | trill | 1 |
| 431 | breeding birds | 1 |
| 432 | antiphony song | 1 |
433 rows × 2 columns
# Compter la fréquence de chaque mot dans l'ensemble des données
individual_words = Counter(clean_word(word) for sublist in cleaned_words for word in sublist.split())
# Convertir le compteur en DataFrame et trier par fréquence décroissante
individual_word_counts_df = pd.DataFrame(individual_words.items(), columns=['Word', 'Frequency']).sort_values(by='Frequency', ascending=False).reset_index(drop=True)
individual_word_counts_df
| Word | Frequency | |
|---|---|---|
| 0 | call | 15489 |
| 1 | song | 5891 |
| 2 | flight | 5119 |
| 3 | uncertain | 1995 |
| 4 | male | 1926 |
| ... | ... | ... |
| 484 | afternoon | 1 |
| 485 | mid | 1 |
| 486 | calljuvenile | 1 |
| 487 | conversational | 1 |
| 488 | antiphony | 1 |
489 rows × 2 columns
# Exclure les chaînes vides et obtenir le top 10
top_10_words = individual_word_counts_df[individual_word_counts_df['Word'] != ''].head(10)
top_10_words
| Word | Frequency | |
|---|---|---|
| 0 | call | 15489 |
| 1 | song | 5891 |
| 2 | flight | 5119 |
| 3 | uncertain | 1995 |
| 4 | male | 1926 |
| 5 | nocturnal | 1657 |
| 6 | adult | 1614 |
| 7 | sex | 1372 |
| 8 | alarm | 929 |
| 9 | female | 713 |
Curiosité à propos de flight : cri en vol ou bruit des ailes ?
# Fonction pour vérifier si 'flight' est dans un des éléments de la liste
def contains_flight(types_list):
return any('flight' in item for item in types_list)
# Filtrer pour obtenir les lignes où la colonne 'contains_flight' est True
filtered_data = birds_df[birds_df['type'].apply(contains_flight)]
filtered_data.head()
| TAXON_ORDER | CATEGORY | SPECIES_CODE | PRIMARY_COM_NAME | SCI_NAME | ORDER1 | FAMILY | primary_label | secondary_labels | type | latitude | longitude | scientific_name | common_name | author | license | rating | url | filename | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 233 | species | lewduc1 | Lesser Whistling-Duck | Dendrocygna javanica | Anseriformes | Anatidae (Ducks, Geese, and Waterfowl) | lewduc1 | [] | [flight call, song] | 20.2609 | 100.0315 | Dendrocygna javanica | Lesser Whistling-Duck | Greg Irving | Creative Commons Attribution-NonCommercial-Sha... | 4.0 | https://www.xeno-canto.org/169762 | lewduc1/XC169762.ogg |
| 5 | 233 | species | lewduc1 | Lesser Whistling-Duck | Dendrocygna javanica | Anseriformes | Anatidae (Ducks, Geese, and Waterfowl) | lewduc1 | [] | [call, flight call] | 7.9610 | 80.7590 | Dendrocygna javanica | Lesser Whistling-Duck | Mike Nelson | Creative Commons Attribution-NonCommercial-Sha... | 4.0 | https://www.xeno-canto.org/207641 | lewduc1/XC207641.ogg |
| 8 | 233 | species | lewduc1 | Lesser Whistling-Duck | Dendrocygna javanica | Anseriformes | Anatidae (Ducks, Geese, and Waterfowl) | lewduc1 | [] | [flight call] | 28.6958 | 80.9467 | Dendrocygna javanica | Lesser Whistling-Duck | Benj Smelt+Hiru (BCN) | Creative Commons Attribution-NonCommercial-Sha... | 4.0 | https://www.xeno-canto.org/261506 | lewduc1/XC261506.ogg |
| 9 | 233 | species | lewduc1 | Lesser Whistling-Duck | Dendrocygna javanica | Anseriformes | Anatidae (Ducks, Geese, and Waterfowl) | lewduc1 | [] | [flight call] | 10.1691 | 76.4396 | Dendrocygna javanica | Lesser Whistling-Duck | Dilip KG | Creative Commons Attribution-NonCommercial-Sha... | 4.0 | https://www.xeno-canto.org/276848 | lewduc1/XC276848.ogg |
| 10 | 233 | species | lewduc1 | Lesser Whistling-Duck | Dendrocygna javanica | Anseriformes | Anatidae (Ducks, Geese, and Waterfowl) | lewduc1 | [] | [flight call] | 10.1691 | 76.4396 | Dendrocygna javanica | Lesser Whistling-Duck | Dilip KG | Creative Commons Attribution-NonCommercial-Sha... | 3.0 | https://www.xeno-canto.org/276849 | lewduc1/XC276849.ogg |
# Exemple de flight call
# Lesser Whistling-Duck : Dendrocygne siffleur
bird3='train/lewduc1/XC37524.ogg'
audio_spectro(bird3)
# Filtrer les chaînes qui contiennent le mot "islands", insensible à la casse
filtered_skies = [s for s in unique_items_list if "islands" in s.lower()]
print(filtered_skies)
[]
Dommage une information de ce type aurait été la bienvenue.
La colonne rating¶
birds_df[birds_df['rating']<=1.5]
| TAXON_ORDER | CATEGORY | SPECIES_CODE | PRIMARY_COM_NAME | SCI_NAME | ORDER1 | FAMILY | primary_label | secondary_labels | type | latitude | longitude | scientific_name | common_name | author | license | rating | url | filename | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 42 | 425 | species | gargan | Garganey | Spatula querquedula | Anseriformes | Anatidae (Ducks, Geese, and Waterfowl) | gargan | [] | [flight call] | 50.1263 | 59.0844 | Spatula querquedula | Garganey | Albert Lastukhin | Creative Commons Attribution-NonCommercial-Sha... | 1.5 | https://www.xeno-canto.org/182411 | gargan/XC182411.ogg |
| 63 | 425 | species | gargan | Garganey | Spatula querquedula | Anseriformes | Anatidae (Ducks, Geese, and Waterfowl) | gargan | [] | [call, song] | 50.3654 | 8.8852 | Spatula querquedula | Garganey | brickegickel | Creative Commons Attribution-NonCommercial-Sha... | 1.5 | https://www.xeno-canto.org/361394 | gargan/XC361394.ogg |
| 77 | 425 | species | gargan | Garganey | Spatula querquedula | Anseriformes | Anatidae (Ducks, Geese, and Waterfowl) | gargan | [] | [call, male] | 63.1874 | 75.3780 | Spatula querquedula | Garganey | Albert Lastukhin | Creative Commons Attribution-NonCommercial-Sha... | 1.5 | https://www.xeno-canto.org/431303 | gargan/XC431303.ogg |
| 111 | 425 | species | gargan | Garganey | Spatula querquedula | Anseriformes | Anatidae (Ducks, Geese, and Waterfowl) | gargan | [] | [male, song] | 60.2409 | 25.2100 | Spatula querquedula | Garganey | Hannu Varkki | Creative Commons Attribution-NonCommercial-Sha... | 1.5 | https://www.xeno-canto.org/642509 | gargan/XC642509.ogg |
| 113 | 425 | species | gargan | Garganey | Spatula querquedula | Anseriformes | Anatidae (Ducks, Geese, and Waterfowl) | gargan | [] | [flight call] | 52.3240 | 10.4527 | Spatula querquedula | Garganey | Sven Kransel | Creative Commons Attribution-NonCommercial-Sha... | 0.5 | https://www.xeno-canto.org/643885 | gargan/XC643885.ogg |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 24294 | 31096 | species | comros | Common Rosefinch | Carpodacus erythrinus | Passeriformes | Fringillidae (Finches, Euphonias, and Allies) | comros | [] | [male, song] | 52.0778 | 4.2361 | Carpodacus erythrinus | Common Rosefinch | Gerjon Gelling | Creative Commons Attribution-NonCommercial-Sha... | 0.0 | https://www.xeno-canto.org/563057 | comros/XC563057.ogg |
| 24295 | 31096 | species | comros | Common Rosefinch | Carpodacus erythrinus | Passeriformes | Fringillidae (Finches, Euphonias, and Allies) | comros | [] | [song] | 49.5143 | 17.3214 | Carpodacus erythrinus | Common Rosefinch | Igor Uřinovský | Creative Commons Attribution-NonCommercial-Sha... | 0.0 | https://www.xeno-canto.org/563477 | comros/XC563477.ogg |
| 24320 | 31096 | species | comros | Common Rosefinch | Carpodacus erythrinus | Passeriformes | Fringillidae (Finches, Euphonias, and Allies) | comros | [] | [song] | 58.3971 | 26.5377 | Carpodacus erythrinus | Common Rosefinch | Stanislas Wroza | Creative Commons Attribution-NonCommercial-Sha... | 0.0 | https://www.xeno-canto.org/575049 | comros/XC575049.ogg |
| 24322 | 31096 | species | comros | Common Rosefinch | Carpodacus erythrinus | Passeriformes | Fringillidae (Finches, Euphonias, and Allies) | comros | [] | [song] | 46.2581 | 10.5807 | Carpodacus erythrinus | Common Rosefinch | Sergio Mazzotti | Creative Commons Attribution-NonCommercial-Sha... | 0.0 | https://www.xeno-canto.org/575827 | comros/XC575827.ogg |
| 24349 | 31096 | species | comros | Common Rosefinch | Carpodacus erythrinus | Passeriformes | Fringillidae (Finches, Euphonias, and Allies) | comros | [] | [song] | 58.4213 | 26.2352 | Carpodacus erythrinus | Common Rosefinch | Uku Paal | Creative Commons Attribution-NonCommercial-Sha... | 1.0 | https://www.xeno-canto.org/653240 | comros/XC653240.ogg |
1127 rows × 19 columns
# Exemple de rating a 1.5
# Garganey : Sarcelle d'été
bird4='train/gargan/XC361394.ogg'
audio_spectro(bird4)
On reconnait une sorte de canard donc on fait le pari qu'on peut extraitre des motifs suffisamment fidèles. On conserve les rating 1.5
# Exemple de rating a 1
# Common Rosefinch : Roselin cramoisi
bird5='train/comros/XC653240.ogg'
audio_spectro(bird5)
Les motifs sont facilement repérables à l'oeil et on les entend facilement. Seul problème : leur intensité va probablement exiger qu'on module le seuil de repérages des motifs suivant le rating.
On va supprimer de qualité inférieure ou égale à 0,5
birds_df = birds_df[birds_df['rating']>0.5]
# Point d'étape
summarize_dataframe(birds_df)
| Column Name | Missing Data (%) | Number |
|---|---|---|
| Total Rows | - | 23815 |
| Total Columns | - | 19 |
| latitude | 1.24 | 295 |
| longitude | 1.24 | 295 |
Répartition sur la planète¶
fig = px.scatter_mapbox(birds_df, lat='latitude', lon='longitude', color='ORDER1',
hover_name='common_name', hover_data=['latitude', 'longitude'],
title='Lieux des enregistrements',
zoom=1, height=600, template='ggplot2')
fig.update_layout(
mapbox_style="white-bg",
mapbox_layers=[
{
"below": 'traces',
"sourcetype": "raster",
"sourceattribution": "© OpenStreetMap contributors",
"source": [
"https://tile.openstreetmap.org/{z}/{x}/{y}.png"
]
}
])
fig.show()
Filtre sur les Ghâts occidentaux¶
# Chargement des données GeoJSON
gdf = gpd.read_file('Western_Ghats/WG.geojson')
# Vérification des données pour sélection des colonnes pertinentes si nécessaire
print(gdf.head())
NAME TYPE NAME_TYPE Color latitude \ 0 Western Ghats hotspot_area Western Ghats_hotspot_area 2 19.101145 longitude geometry 0 77.101145 MULTIPOLYGON (((72.75904 19.93054, 72.75739 19...
# On s'assure que le CRS est approprié pour la visualisation (WGS84 - EPSG:4326)
gdf = gdf.to_crs(epsg=4326)
# Création d'une carte choroplèthe en utilisant la colonne 'TYPE' pour la couleur
fig = px.choropleth(gdf, geojson=gdf.geometry, locations=gdf.index, color="TYPE")
# Mettre à jour les paramètres géographiques pour afficher uniquement la zone pertinente
fig.update_geos(
fitbounds="locations",
visible=True, # Changer à 'True' pour rendre visible les frontières et autres informations géographiques
showcountries=True, # Affiche les frontières des pays
countrycolor="RebeccaPurple" # Couleur des frontières des pays, modifiable selon vos préférences
)
# Ajuster la vue de la carte pour se concentrer sur les Ghâts occidentaux
fig.update_layout(
margin={"r":0,"t":0,"l":0,"b":0},
geo=dict(
lonaxis_range=[72.5, 78], # Longitude minimale et maximale
lataxis_range=[8, 21], # Latitude minimale et maximale,
showframe=True, # Afficher un cadre autour de la carte
showcoastlines=True, # Afficher les lignes côtières
coastlinecolor="Blue", # Couleur des lignes côtières
projection_scale=5 # Ajuster le niveau de zoom si nécessaire
)
)
# Afficher la figure
fig.show()
Création du filtre de la zone des Ghâts
# Convertir le DataFrame en GeoDataFrame
gdf_birds_df = gpd.GeoDataFrame(birds_df, geometry=gpd.points_from_xy(birds_df.longitude, birds_df.latitude))
# Assurer que les données sont en WGS84 (EPSG:4326)
gdf_birds_df.crs = 'epsg:4326'
# Charger le fichier GeoJSON
gdf_polygons_wg = gpd.read_file('Western_Ghats/WG.geojson')
gdf_polygons_wg = gdf_polygons_wg.to_crs('epsg:4326') # Assurez-vous que le CRS correspond à celui des points
# Filtrer les points qui sont à l'intérieur des polygones des Ghâts occidentaux
points_within_wg = gdf_birds_df[gdf_birds_df.geometry.within(gdf_polygons_wg.unary_union)]
# Visualiser avec Plotly
fig = px.scatter_mapbox(points_within_wg,
lat=points_within_wg.geometry.y,
lon=points_within_wg.geometry.x,
hover_name='common_name', # Remplacez 'some_column' par le nom de colonne pertinent
mapbox_style="open-street-map",
zoom=3, center={"lat": 14.5, "lon": 75.5})
fig.show()
points_within_wg.info()
<class 'geopandas.geodataframe.GeoDataFrame'> Index: 1261 entries, 218 to 23919 Data columns (total 20 columns): # Column Non-Null Count Dtype --- ------ -------------- ----- 0 TAXON_ORDER 1261 non-null int64 1 CATEGORY 1261 non-null object 2 SPECIES_CODE 1261 non-null object 3 PRIMARY_COM_NAME 1261 non-null object 4 SCI_NAME 1261 non-null object 5 ORDER1 1261 non-null object 6 FAMILY 1261 non-null object 7 primary_label 1261 non-null object 8 secondary_labels 1261 non-null object 9 type 1261 non-null object 10 latitude 1261 non-null float64 11 longitude 1261 non-null float64 12 scientific_name 1261 non-null object 13 common_name 1261 non-null object 14 author 1261 non-null object 15 license 1261 non-null object 16 rating 1261 non-null float64 17 url 1261 non-null object 18 filename 1261 non-null object 19 geometry 1261 non-null geometry dtypes: float64(3), geometry(1), int64(1), object(15) memory usage: 206.9+ KB
# Création d'un polygone unique s'il y a plusieurs polygones dans le GeoJSON
polygon_wg = gdf_polygons_wg.unary_union
# Filtrer pour obtenir seulement les points à l'intérieur du polygone des Ghâts occidentaux
points_within_wg = gdf_birds_df[gdf_birds_df.geometry.within(polygon_wg)]
# points_within contient maintenant seulement les points qui sont à l'intérieur des Ghâts occidentaux
points_within_wg.info()
# Déterminer si chaque point est à l'intérieur du polygone ghats
gdf_birds_df['in_ghats'] = gdf_birds_df.geometry.within(polygon_wg)
# Convertir les valeurs booléennes True/False en 1/0
# et ajouter la colonne booléenne au DataFrame principal
birds_df['in_ghats'] = gdf_birds_df['in_ghats'].astype(int)
<class 'geopandas.geodataframe.GeoDataFrame'> Index: 1261 entries, 218 to 23919 Data columns (total 20 columns): # Column Non-Null Count Dtype --- ------ -------------- ----- 0 TAXON_ORDER 1261 non-null int64 1 CATEGORY 1261 non-null object 2 SPECIES_CODE 1261 non-null object 3 PRIMARY_COM_NAME 1261 non-null object 4 SCI_NAME 1261 non-null object 5 ORDER1 1261 non-null object 6 FAMILY 1261 non-null object 7 primary_label 1261 non-null object 8 secondary_labels 1261 non-null object 9 type 1261 non-null object 10 latitude 1261 non-null float64 11 longitude 1261 non-null float64 12 scientific_name 1261 non-null object 13 common_name 1261 non-null object 14 author 1261 non-null object 15 license 1261 non-null object 16 rating 1261 non-null float64 17 url 1261 non-null object 18 filename 1261 non-null object 19 geometry 1261 non-null geometry dtypes: float64(3), geometry(1), int64(1), object(15) memory usage: 206.9+ KB
# Regard sur les 3 colonnes concernées
print(birds_df[['latitude', 'longitude', 'in_ghats']])
latitude longitude in_ghats 0 13.9144 104.5538 0 1 20.2609 100.0315 0 2 6.2917 81.2685 0 3 6.2917 81.2685 0 4 6.1702 81.2054 0 ... ... ... ... 24454 48.0372 -4.8529 0 24455 48.0372 -4.8529 0 24456 56.2381 16.4529 0 24457 56.0167 15.7833 0 24458 53.1080 14.3437 0 [23815 rows x 3 columns]
# Point d'étape
summarize_dataframe(birds_df)
| Column Name | Missing Data (%) | Number |
|---|---|---|
| Total Rows | - | 23815 |
| Total Columns | - | 20 |
| latitude | 1.24 | 295 |
| longitude | 1.24 | 295 |
birds_df.in_ghats.value_counts()
in_ghats 0 22554 1 1261 Name: count, dtype: int64
Sri Lanka¶
Les deux régions abritent un assemblage riche de plantes endémiques, de reptiles et d’amphibiens. Les Ghâts occidentaux, connus localement sous le nom Hills Sahyadri, sont formés par les plaines de Malabar et la chaîne de montagne parallèle à la côte ouest de l’Inde. Le Sri Lanka est une île séparée du continent, du sud de l’Inde, par les 20 mètres de profondeur du détroit de Palk.
Ces deux régions jouent un rôle important dans l’interception des vents de la mousson du sud-ouest. La grande variabilité de la pluviosité dans les Ghâts occidentaux, couplée avec la géographie complexe de la région, produit une grande variété de types de végétation1.
Cependant, il est important de noter que ces régions sont menacées par la pression énorme de la population et ont été considérablement impactées par la demande en bois et en terres agricoles.
# Charger le fichier GeoJSON
gdf_polygons_sl = gpd.read_file('Western_Ghats/SL.geojson')
gdf_polygons_sl = gdf_polygons_sl.to_crs('epsg:4326') # Assurez-vous que le CRS correspond à celui des points
# Filtrer les points qui sont à l'intérieur des polygones du Sri Lanka
points_within_sl = gdf_birds_df[gdf_birds_df.geometry.within(gdf_polygons_sl.unary_union)]
# Visualiser avec Plotly
fig = px.scatter_mapbox(points_within_sl,
lat=points_within_sl.geometry.y,
lon=points_within_sl.geometry.x,
hover_name='common_name', # Remplacez 'some_column' par le nom de colonne pertinent
mapbox_style="open-street-map",
zoom=3, center={"lat": 14.5, "lon": 75.5})
fig.show()
points_within_sl.info()
<class 'geopandas.geodataframe.GeoDataFrame'> Index: 385 entries, 2 to 23950 Data columns (total 21 columns): # Column Non-Null Count Dtype --- ------ -------------- ----- 0 TAXON_ORDER 385 non-null int64 1 CATEGORY 385 non-null object 2 SPECIES_CODE 385 non-null object 3 PRIMARY_COM_NAME 385 non-null object 4 SCI_NAME 385 non-null object 5 ORDER1 385 non-null object 6 FAMILY 385 non-null object 7 primary_label 385 non-null object 8 secondary_labels 385 non-null object 9 type 385 non-null object 10 latitude 385 non-null float64 11 longitude 385 non-null float64 12 scientific_name 385 non-null object 13 common_name 385 non-null object 14 author 385 non-null object 15 license 385 non-null object 16 rating 385 non-null float64 17 url 385 non-null object 18 filename 385 non-null object 19 geometry 385 non-null geometry 20 in_ghats 385 non-null bool dtypes: bool(1), float64(3), geometry(1), int64(1), object(15) memory usage: 63.5+ KB
# Création d'un polygone unique s'il y a plusieurs polygones dans le GeoJSON
polygon_sl = gdf_polygons_sl.unary_union
# Filtrer pour obtenir seulement les points à l'intérieur du polygone des Ghâts occidentaux
points_within_sl = gdf_birds_df[gdf_birds_df.geometry.within(polygon_sl)]
# points_within contient maintenant seulement les points qui sont à l'intérieur des Ghâts occidentaux
points_within_sl.info()
<class 'geopandas.geodataframe.GeoDataFrame'> Index: 385 entries, 2 to 23950 Data columns (total 21 columns): # Column Non-Null Count Dtype --- ------ -------------- ----- 0 TAXON_ORDER 385 non-null int64 1 CATEGORY 385 non-null object 2 SPECIES_CODE 385 non-null object 3 PRIMARY_COM_NAME 385 non-null object 4 SCI_NAME 385 non-null object 5 ORDER1 385 non-null object 6 FAMILY 385 non-null object 7 primary_label 385 non-null object 8 secondary_labels 385 non-null object 9 type 385 non-null object 10 latitude 385 non-null float64 11 longitude 385 non-null float64 12 scientific_name 385 non-null object 13 common_name 385 non-null object 14 author 385 non-null object 15 license 385 non-null object 16 rating 385 non-null float64 17 url 385 non-null object 18 filename 385 non-null object 19 geometry 385 non-null geometry 20 in_ghats 385 non-null bool dtypes: bool(1), float64(3), geometry(1), int64(1), object(15) memory usage: 63.5+ KB
# Déterminer si chaque point est à l'intérieur du polygone sri lanka
gdf_birds_df['in_sl'] = gdf_birds_df.geometry.within(polygon_sl)
# Ajouter la colonne booléenne au DataFrame original
# Convertir les valeurs booléennes True/False en 1/0
birds_df['in_sl'] = gdf_birds_df['in_sl'].astype(int)
# Vérifier les résultats
print(birds_df[['latitude', 'longitude', 'in_ghats', 'in_sl']])
latitude longitude in_ghats in_sl 0 13.9144 104.5538 0 0 1 20.2609 100.0315 0 0 2 6.2917 81.2685 0 1 3 6.2917 81.2685 0 1 4 6.1702 81.2054 0 1 ... ... ... ... ... 24454 48.0372 -4.8529 0 0 24455 48.0372 -4.8529 0 0 24456 56.2381 16.4529 0 0 24457 56.0167 15.7833 0 0 24458 53.1080 14.3437 0 0 [23815 rows x 4 columns]
birds_df.describe()
| TAXON_ORDER | latitude | longitude | rating | in_ghats | in_sl | |
|---|---|---|---|---|---|---|
| count | 23815.000000 | 23520.000000 | 23520.000000 | 23815.000000 | 23815.000000 | 23815.000000 |
| mean | 14171.450724 | 32.510576 | 43.878847 | 3.946567 | 0.052950 | 0.016166 |
| std | 9722.846386 | 19.446732 | 50.242738 | 0.916893 | 0.223938 | 0.126117 |
| min | 233.000000 | -43.524000 | -171.765400 | 1.000000 | 0.000000 | 0.000000 |
| 25% | 6028.000000 | 17.035550 | 2.796700 | 3.500000 | 0.000000 | 0.000000 |
| 50% | 9389.000000 | 37.137900 | 27.174450 | 4.000000 | 0.000000 | 0.000000 |
| 75% | 23291.000000 | 49.205800 | 85.366750 | 5.000000 | 0.000000 | 0.000000 |
| max | 31096.000000 | 71.964000 | 177.447800 | 5.000000 | 1.000000 | 1.000000 |
birds_df.in_ghats.value_counts()
in_ghats 0 22554 1 1261 Name: count, dtype: int64
birds_df.in_sl.value_counts()
in_sl 0 23430 1 385 Name: count, dtype: int64
# Point d'étape
summarize_dataframe(birds_df)
| Column Name | Missing Data (%) | Number |
|---|---|---|
| Total Rows | - | 23815 |
| Total Columns | - | 21 |
| latitude | 1.24 | 295 |
| longitude | 1.24 | 295 |
Skies islands¶
Les îles célestes de shola se trouvent au sud des collines de Baba Budan dans le Karnataka, à environ 15 degrés nord. Il existe de nombreuses îles isolées ; certaines dans le Karnataka et le nord du Kerala sont assez petites, mesurant seulement environ 10 à 20 km², tandis que d'autres îles plus grandes comme les Nilgiris et les collines d'Anamalai-Palani ont une superficie d'environ 1500 km². Plus au sud, les « high-wavies » (Meghamalai) et les collines Ashambu (Agasthyamalai) sont à nouveau beaucoup plus petites.
De ce fait il sera difficile de créer un filtre sur les skies islands. On le fera "manuellement" à l'aide du rapport Power Bi ci-après"
Le rapport Power Bi¶
from IPython.display import display, HTML
# Ouverture du dashboard Power BI
html_output = "<a href='https://app.powerbi.com/view?r=eyJrIjoiMmMzYTVkZTUtODM1MC00OGRhLWJlZDMtZDg1Mzc4MTg4ZmNkIiwidCI6ImMyZGM0MmRhLTVjOTktNDRiZS05MjNkLTU4YTk4NTZhYTQ0ZSJ9' target='_blank'>Aller vers le dashboard</a>"
display(HTML(html_output))
Les oiseaux endémiques¶
Notre définition adoptée ici : un oiseau endémique aux Ghâts occidentaux sera un oiseau présent dans cette zone géographique et pas ailleurs
from IPython.display import display, HTML
# Filtrer les observations dans les Ghâts
in_ghats = birds_df[birds_df['in_ghats'] == 1]
not_in_ghats = birds_df[birds_df['in_ghats'] == 0]
# Trouver les noms scientifiques uniques à la région des Ghâts
unique_in_ghats = set(in_ghats['scientific_name']) - set(not_in_ghats['scientific_name'])
# Créer et afficher des liens cliquables pour chaque nom scientifique unique
html_output = "<ul>"
for name in unique_in_ghats:
url = in_ghats[in_ghats['scientific_name'] == name]['url'].iloc[0] # Assurez-vous que chaque nom a une URL
html_output += f"<li><a href='{url}' target='_blank'>{name}</a></li>"
html_output += "</ul>"
display(HTML(html_output))
Par curiosité, au Sri Lanka :
# Filtrer les observations dans le Sri Lanka
in_sl = birds_df[birds_df['in_sl'] == 1]
not_in_sl = birds_df[birds_df['in_sl'] == 0]
# Trouver les noms scientifiques uniques au Sri Lanka
unique_in_sl = set(in_sl['scientific_name']) - set(not_in_sl['scientific_name'])
html_output = "<ul>"
for name in unique_in_sl:
url = in_sl[in_sl['scientific_name'] == name]['url'].iloc[0] # Assurez-vous que chaque nom a une URL
html_output += f"<li><a href='{url}' target='_blank'>{name}</a></li>"
html_output += "</ul>"
display(HTML(html_output))
Aucun oiseau endémique au Sri Lanka
# Convertir l'ensemble en liste
endemic = list(unique_in_ghats)
endemic
['Ocyceros griseus', 'Eumyias albicaudatus', 'Dendrocitta leucogastra', 'Montecincla fairbanki', 'Tephrodornis sylvicola', 'Pterorhinus delesserti', 'Cyornis pallidipes', 'Psilopogon malabaricus']
# Ajouter la colonne booléenne au DataFrame principal
# Convertir les valeurs booléennes True/False en 1/0
birds_df['is_endemic_ghats'] = birds_df["scientific_name"].isin(endemic).astype(int)
birds_df.describe()
| TAXON_ORDER | latitude | longitude | rating | in_ghats | in_sl | is_endemic_ghats | |
|---|---|---|---|---|---|---|---|
| count | 23815.000000 | 23520.000000 | 23520.000000 | 23815.000000 | 23815.000000 | 23815.000000 | 23815.000000 |
| mean | 14171.450724 | 32.510576 | 43.878847 | 3.946567 | 0.052950 | 0.016166 | 0.004157 |
| std | 9722.846386 | 19.446732 | 50.242738 | 0.916893 | 0.223938 | 0.126117 | 0.064342 |
| min | 233.000000 | -43.524000 | -171.765400 | 1.000000 | 0.000000 | 0.000000 | 0.000000 |
| 25% | 6028.000000 | 17.035550 | 2.796700 | 3.500000 | 0.000000 | 0.000000 | 0.000000 |
| 50% | 9389.000000 | 37.137900 | 27.174450 | 4.000000 | 0.000000 | 0.000000 | 0.000000 |
| 75% | 23291.000000 | 49.205800 | 85.366750 | 5.000000 | 0.000000 | 0.000000 | 0.000000 |
| max | 31096.000000 | 71.964000 | 177.447800 | 5.000000 | 1.000000 | 1.000000 | 1.000000 |
birds_df.is_endemic_ghats.value_counts()
is_endemic_ghats 0 23716 1 99 Name: count, dtype: int64
# Point d'étape
summarize_dataframe(birds_df)
| Column Name | Missing Data (%) | Number |
|---|---|---|
| Total Rows | - | 23815 |
| Total Columns | - | 22 |
| latitude | 1.24 | 295 |
| longitude | 1.24 | 295 |
Les oiseaux nocturnes¶

# Filtrer les chaînes qui contiennent le mot "nocturnal", insensible à la casse
filtered_nocturnal = [s for s in unique_items_list if "nocturnal" in s.lower()]
print(filtered_nocturnal)
['nocturnal song', 'nocturnal migration', 'nocturnal flightcall', 'Nocturnal roosting site', 'nocturnal', 'Nocturnal rooting site', 'nocturnal call', 'nocturnal flight call', 'nocturnal flight call', 'Nocturnal', 'Nocturnal call', 'nocturnal migrant', 'Nocturnal flight call.', 'Nocturnal flight call', 'Nocturnal Flight Call']
# Ajout de crochets autour de chaque élément de la liste
filtered_nocturnal = [[item] for item in filtered_nocturnal]
print(filtered_nocturnal)
birds_df_nocturnal=birds_df[birds_df['type'].isin(filtered_nocturnal)]
[['nocturnal song'], ['nocturnal migration'], ['nocturnal flightcall'], ['Nocturnal roosting site'], ['nocturnal'], ['Nocturnal rooting site'], ['nocturnal call'], ['nocturnal flight call'], ['nocturnal flight call'], ['Nocturnal'], ['Nocturnal call'], ['nocturnal migrant'], ['Nocturnal flight call.'], ['Nocturnal flight call'], ['Nocturnal Flight Call']]
birds_df_nocturnal.info()
<class 'pandas.core.frame.DataFrame'> Index: 918 entries, 93 to 23713 Data columns (total 22 columns): # Column Non-Null Count Dtype --- ------ -------------- ----- 0 TAXON_ORDER 918 non-null int64 1 CATEGORY 918 non-null object 2 SPECIES_CODE 918 non-null object 3 PRIMARY_COM_NAME 918 non-null object 4 SCI_NAME 918 non-null object 5 ORDER1 918 non-null object 6 FAMILY 918 non-null object 7 primary_label 918 non-null object 8 secondary_labels 918 non-null object 9 type 918 non-null object 10 latitude 916 non-null float64 11 longitude 916 non-null float64 12 scientific_name 918 non-null object 13 common_name 918 non-null object 14 author 918 non-null object 15 license 918 non-null object 16 rating 918 non-null float64 17 url 918 non-null object 18 filename 918 non-null object 19 in_ghats 918 non-null int64 20 in_sl 918 non-null int64 21 is_endemic_ghats 918 non-null int64 dtypes: float64(3), int64(4), object(15) memory usage: 165.0+ KB
birds_df[birds_df['type'].isin(filtered_nocturnal)].FAMILY.unique()
array(['Anatidae (Ducks, Geese, and Waterfowl)', 'Podicipedidae (Grebes)',
'Rallidae (Rails, Gallinules, and Coots)',
'Recurvirostridae (Stilts and Avocets)',
'Charadriidae (Plovers and Lapwings)',
'Scolopacidae (Sandpipers and Allies)',
'Laridae (Gulls, Terns, and Skimmers)',
'Ardeidae (Herons, Egrets, and Bitterns)',
'Alcedinidae (Kingfishers)', 'Psittaculidae (Old World Parrots)',
'Hirundinidae (Swallows)', 'Motacillidae (Wagtails and Pipits)'],
dtype=object)
- Strigidae : Cette famille est largement représentée en Inde avec des espèces comme la chouette hulotte, la chouette de l'Himalaya, et la chouette effraie. Ces oiseaux sont présents dans divers habitats, des forêts aux zones urbaines.
- Tytonidae : L'effraie des clochers est le représentant le plus connu de cette famille en Inde. On la trouve souvent près des habitats humains et dans les fermes, où elle chasse les rongeurs la nuit.
- Caprimulgidae : Les engoulevents, tels que l'engoulevent indien et l'engoulevent sykes, sont répandus à travers le sous-continent. Ils sont connus pour leur plumage cryptique qui les rend difficiles à repérer lorsqu'ils sont posés au sol pendant la journée.
- Podargidae : Bien que moins communs, certains "frogmouths" tels que le frogmouth de Ceylan peuvent être trouvés dans les régions boisées d'Inde, surtout dans le sud du pays.
- Ces familles montrent une adaptation remarquable à divers environnements, de la forêt dense aux montagnes et aux zones urbaines, jouant des rôles écologiques importants, notamment dans le contrôle des populations d'insectes et de petits mammifères.
nocturnal = ['Strigidae','Tytonidae','Caprimulgidae','Podargidae']
# Création d'une expression régulière pour matcher toute famille dans la liste nocturnal
import re
pattern = '|'.join(re.escape(family) for family in nocturnal)
# Filtrage du DataFrame
filtered_df = birds_df_nocturnal[birds_df_nocturnal['FAMILY'].str.contains(pattern)]
filtered_df.info()
<class 'pandas.core.frame.DataFrame'> Index: 0 entries Data columns (total 22 columns): # Column Non-Null Count Dtype --- ------ -------------- ----- 0 TAXON_ORDER 0 non-null int64 1 CATEGORY 0 non-null object 2 SPECIES_CODE 0 non-null object 3 PRIMARY_COM_NAME 0 non-null object 4 SCI_NAME 0 non-null object 5 ORDER1 0 non-null object 6 FAMILY 0 non-null object 7 primary_label 0 non-null object 8 secondary_labels 0 non-null object 9 type 0 non-null object 10 latitude 0 non-null float64 11 longitude 0 non-null float64 12 scientific_name 0 non-null object 13 common_name 0 non-null object 14 author 0 non-null object 15 license 0 non-null object 16 rating 0 non-null float64 17 url 0 non-null object 18 filename 0 non-null object 19 in_ghats 0 non-null int64 20 in_sl 0 non-null int64 21 is_endemic_ghats 0 non-null int64 dtypes: float64(3), int64(4), object(15) memory usage: 0.0+ bytes
On constate qu'aucun enregistrement dont la colonne type inclut au moins une fois le mot 'nocturnal' ne concerne un oiseau appartenant à une de ces 4 familles. Par prudence on va indiquer que ces 4 familles concernent des oiseaux nocturnes.
birds_df[birds_df['FAMILY'].isin(nocturnal).astype(int)==1]
| TAXON_ORDER | CATEGORY | SPECIES_CODE | PRIMARY_COM_NAME | SCI_NAME | ORDER1 | FAMILY | primary_label | secondary_labels | type | ... | scientific_name | common_name | author | license | rating | url | filename | in_ghats | in_sl | is_endemic_ghats |
|---|
0 rows × 22 columns
Aucune des familles classiques d'oiseaux nocturnes n'est présente dans le jeu de données. On laisse quand même apparaître cette condition dans la cellule en cas d'ajout futur de données concernant ces familles.
birds_df['is_nocturnal'] = ((birds_df['type'].isin(filtered_nocturnal)) | (birds_df['FAMILY'].isin(nocturnal))).astype(int)
birds_df.is_nocturnal.value_counts()
is_nocturnal 0 22897 1 918 Name: count, dtype: int64
# Point d'étape
summarize_dataframe(birds_df)
| Column Name | Missing Data (%) | Number |
|---|---|---|
| Total Rows | - | 23815 |
| Total Columns | - | 23 |
| latitude | 1.24 | 295 |
| longitude | 1.24 | 295 |
Les oiseaux en danger¶
Taille et déclin de la population : On mesure le nombre total d'individus adultes dans la population, ainsi que le taux et la rapidité du déclin de cette population. Une espèce est souvent considérée en danger si sa population est inférieure à un seuil critique ou si elle montre un déclin rapide.
Aire de répartition géographique : La taille de l'aire de répartition est un critère important. Une espèce peut être classée en danger si son aire de répartition est très petite ou fragmentée, ou si elle subit une réduction continue de cette aire.
Taille de la population ou aire de répartition extrêmement réduite : Des critères spécifiques existent pour les espèces dont la population est très petite ou dont l'aire de répartition est extrêmement limitée, ce qui les rend vulnérables à des événements catastrophiques ou des changements environnementaux.
Probabilité d'extinction : Des modélisations statistiques sont utilisées pour estimer la probabilité d'extinction dans la nature, basée sur des facteurs comme la dégradation de l'habitat, les impacts climatiques, la présence de prédateurs ou de maladies, etc.
Facteurs spécifiques : Pour certaines espèces, des facteurs plus spécifiques peuvent être pris en compte, tels que les effets de la consanguinité, les taux de reproduction extrêmement bas, ou d'autres menaces biologiques ou environnementales spécifiques.
Ces critères sont appliqués dans un cadre systématique pour classer les espèces dans différentes catégories de menace, telles que "en danger critique", "en danger", et "vulnérable", chacune avec des définitions et des critères spécifiques.
# Filtrer les données pour inclure uniquement celles où 'ghats' est égal à 1
ghats_data = birds_df[birds_df['in_ghats'] == 1]
# Groupement des données par 'common_name' et agrégation des valeurs min et max de 'latitude' et 'longitude'
range_data = ghats_data.groupby('common_name').agg({
'latitude': ['min', 'max'],
'longitude': ['min', 'max']
}).reset_index()[:2]
range_data.columns = ['common_name', 'min_latitude', 'max_latitude', 'min_longitude', 'max_longitude']
# Déterminer les limites pour le zoom
min_lat = range_data['min_latitude'].min()
max_lat = range_data['max_latitude'].max()
min_lon = range_data['min_longitude'].min()
max_lon = range_data['max_longitude'].max()
# Fonction pour dessiner les bounding boxes sur la carte
def draw_bounding_box(ax, row):
ax.add_patch(plt.Rectangle((row['min_longitude'], row['min_latitude']),
row['max_longitude'] - row['min_longitude'],
row['max_latitude'] - row['min_latitude'],
fill=False, edgecolor='red', lw=2))
ax.text(row['min_longitude'], row['min_latitude'], row['common_name'], verticalalignment='top')
# Création de la figure et de l'axe
fig, ax = plt.subplots(figsize=(10, 6))
world = gpd.read_file(gpd.datasets.get_path('naturalearth_lowres'))
world.plot(ax=ax, color='white', edgecolor='black')
# Appliquer les bounding boxes
range_data.apply(lambda row: draw_bounding_box(ax, row), axis=1)
# Ajuster les limites de la carte pour se concentrer sur les zones d'intérêt
ax.set_xlim(min_lon, max_lon)
ax.set_ylim(min_lat, max_lat)
plt.show()
/var/folders/tz/4wtc6b996hn3rzhm9bbb2ph80000gn/T/ipykernel_4876/2924694588.py:17: FutureWarning: The geopandas.dataset module is deprecated and will be removed in GeoPandas 1.0. You can get the original 'naturalearth_lowres' data from https://www.naturalearthdata.com/downloads/110m-cultural-vectors/.
import geopandas as gpd
import plotly.express as px
import plotly.graph_objects as go
import matplotlib.pyplot as plt
'''# Chargement des données GeoJSON
gdf = gpd.read_file('Western_Ghats/WG.geojson')
# On s'assure que le CRS est approprié pour la visualisation (WGS84 - EPSG:4326)
gdf = gdf.to_crs(epsg=4326)'''
# Renommer la colonne 'TYPE' pour la légende
gdf = gdf.rename(columns={'TYPE': 'Ghats'})
# Création d'une carte choroplèthe en utilisant la colonne 'TYPE' pour la couleur
fig = px.choropleth(gdf, geojson=gdf.geometry, locations=gdf.index, color="Ghats")
# Mettre à jour les paramètres géographiques pour afficher uniquement la zone pertinente
fig.update_geos(
fitbounds="locations",
visible=True, # Rendre visible les frontières et autres informations géographiques
showcountries=True, # Afficher les frontières des pays
countrycolor="RebeccaPurple" # Couleur des frontières des pays
)
# Ajuster la vue de la carte pour se concentrer sur les Ghâts occidentaux
fig.update_layout(
margin={"r":0,"t":0,"l":0,"b":0},
geo=dict(
lonaxis_range=[72.5, 78], # Longitude minimale et maximale
lataxis_range=[8, 21], # Latitude minimale et maximale
showframe=True, # Afficher un cadre autour de la carte
showcoastlines=True, # Afficher les lignes côtières
coastlinecolor="Blue", # Couleur des lignes côtières
projection_scale=5 # Ajuster le niveau de zoom si nécessaire
)
)
'''# Données pour les zones rectangulaires (par exemple)
range_data = pd.DataFrame({
'common_name': ['Zone 1', 'Zone 2'],
'min_latitude': [10, 12],
'max_latitude': [11, 13],
'min_longitude': [75, 76],
'max_longitude': [76, 77]
})'''
# Ajout des zones rectangulaires sur la carte
for _, row in range_data.iterrows():
fig.add_trace(go.Scattergeo(
lon=[row['min_longitude'], row['max_longitude'], row['max_longitude'], row['min_longitude'], row['min_longitude']],
lat=[row['min_latitude'], row['min_latitude'], row['max_latitude'], row['max_latitude'], row['min_latitude']],
mode='lines',
line=dict(width=2, color='red'),
name=row['common_name']
))
# Afficher la figure
fig.show()
# Calculer l'aire de chaque rectangle (en degrés carrés pour la simplicité)
range_data['area'] = (range_data['max_longitude'] - range_data['min_longitude']) * (range_data['max_latitude'] - range_data['min_latitude'])
# Merge range_data back into ghats_data pour ajouter la colonne 'area'
ghats_data = pd.merge(ghats_data, range_data[['common_name', 'area']], on='common_name', how='left')
# Afficher les premières lignes pour vérifier
ghats_data.head()
| TAXON_ORDER | CATEGORY | SPECIES_CODE | PRIMARY_COM_NAME | SCI_NAME | ORDER1 | FAMILY | primary_label | secondary_labels | type | ... | author | license | rating | url | filename | in_ghats | in_sl | is_endemic_ghats | is_nocturnal | area | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 1471 | species | compea | Indian Peafowl | Pavo cristatus | Galliformes | Phasianidae (Pheasants, Grouse, and Allies) | compea | [] | [call] | ... | Andrew Mascarenhas | Creative Commons Attribution-NonCommercial-Sha... | 4.0 | https://www.xeno-canto.org/196740 | compea/XC196740.ogg | 1 | 0 | 0 | 0 | NaN |
| 1 | 1471 | species | compea | Indian Peafowl | Pavo cristatus | Galliformes | Phasianidae (Pheasants, Grouse, and Allies) | compea | [] | [call] | ... | Dilip KG | Creative Commons Attribution-NonCommercial-Sha... | 4.0 | https://www.xeno-canto.org/406117 | compea/XC406117.ogg | 1 | 0 | 0 | 0 | NaN |
| 2 | 1471 | species | compea | Indian Peafowl | Pavo cristatus | Galliformes | Phasianidae (Pheasants, Grouse, and Allies) | compea | [] | [aberrant, adult, call, sex uncertain, song] | ... | Prakash G | Creative Commons Attribution-NonCommercial-Sha... | 3.0 | https://www.xeno-canto.org/565301 | compea/XC565301.ogg | 1 | 0 | 0 | 0 | NaN |
| 3 | 1471 | species | compea | Indian Peafowl | Pavo cristatus | Galliformes | Phasianidae (Pheasants, Grouse, and Allies) | compea | [] | [adult, call, male] | ... | Sreekumar Chirukandoth | Creative Commons Attribution-NonCommercial-Sha... | 5.0 | https://www.xeno-canto.org/644022 | compea/XC644022.ogg | 1 | 0 | 0 | 0 | NaN |
| 4 | 1471 | species | compea | Indian Peafowl | Pavo cristatus | Galliformes | Phasianidae (Pheasants, Grouse, and Allies) | compea | ['grecou1', 'pursun4'] | [adult, call, male] | ... | Sreekumar Chirukandoth | Creative Commons Attribution-NonCommercial-Sha... | 4.5 | https://www.xeno-canto.org/644024 | compea/XC644024.ogg | 1 | 0 | 0 | 0 | NaN |
5 rows × 24 columns
Remarques : Cette méthode de calcul donne une "aire" en degrés carrés, ce qui est utile pour des comparaisons relatives mais ne représente pas une aire physique exacte en termes de kilomètres carrés. Si vous avez besoin de calculer l'aire exacte en kilomètres carrés, il vous faudrait utiliser des fonctions de géolocalisation plus avancées qui tiennent compte de la courbure de la terre et de la variation de la distance couverte par un degré de latitude/longitude selon la latitude.
On pourrait envisager l'utilisation de bibliothèques comme geopandas pour de telles tâches, car elles offrent des outils de manipulation de données géospatiales plus sophistiqués.
# on ne conserve que les oiseaux présents au moins deux fois
filtered_data = ghats_data.groupby('common_name').filter(lambda x: len(x) > 1)
# En général, 1 degré de latitude est approximativement égal à 111 km
ghats_data['area'] = ghats_data['area']*111*111
# On va considérer comme limite de son aire de répartition pour considérer un oiseau en danger à 5000 km2
danger=ghats_data=ghats_data[ghats_data['area'] < 5000]
danger
| TAXON_ORDER | CATEGORY | SPECIES_CODE | PRIMARY_COM_NAME | SCI_NAME | ORDER1 | FAMILY | primary_label | secondary_labels | type | ... | author | license | rating | url | filename | in_ghats | in_sl | is_endemic_ghats | is_nocturnal | area |
|---|
0 rows × 24 columns
# Trier ghats_data par la colonne 'area' en ordre ascendant
sorted_ghats_data = ghats_data.loc[ghats_data['area'] > 0].sort_values(by='area')
# Sélectionner les 10 premières lignes pour obtenir les 10 plus petites aires
top_10_smallest_areas = sorted_ghats_data.head(50)
# Afficher les résultats
print(top_10_smallest_areas[['common_name', 'area']])
Empty DataFrame Columns: [common_name, area] Index: []
# Affichage des résultats uniques
top_10_smallest_areas.drop_duplicates(['common_name', 'area'])[['common_name', 'area']].head(10)
| common_name | area |
|---|
# Extraction des valeurs uniques de la colonne 'common_name' dans un tableau numpy
unique_danger_array = top_10_smallest_areas['common_name'].unique()
# Conversion du tableau numpy en une liste Python
unique_danger = unique_danger_array.tolist()
unique_danger
[]
birds_df['in_danger_ghats'] = birds_df["common_name"].isin(unique_danger).astype(int)
# Point d'étape
summarize_dataframe(birds_df)
| Column Name | Missing Data (%) | Number |
|---|---|---|
| Total Rows | - | 23815 |
| Total Columns | - | 24 |
| latitude | 1.24 | 295 |
| longitude | 1.24 | 295 |
# birds_df.to_csv('birds_audio.csv', index=False)